Introdução
O caminho do xiquimato está estreitamente interligado com os dos aminoácidos aromáticos (L-triptofano, L-fenilalanina, e L-tirosina) e nas plantas terrestres suporta fluxos muito elevados com estimativas da quantidade de carbono fixo que passa pelo caminho variando entre 20 e 50% (Weiss, 1986; Corea et al., 2012; Maeda e Dudareva, 2012). Tem sido dado um enfoque considerável à investigação nesta via desde que os aminoácidos aromáticos não são produzidos pelo homem e pelo gado monogástrico e são, portanto, um componente dietético importante (Tzin e Galili, 2010). Além disso, uma das enzimas do caminho – 5-enolpiruvalshikimate-3-fosfato sintase (EPSP) – é um dos locais alvo de herbicidas mais amplamente empregados (ver, Duke e Powles, 2008). Além disso, como temos descrito recentemente, os metabólitos fenólicos vegetais secundários e seus precursores são sintetizados através da via da biossíntese do xiquimato e seus numerosos pontos de ramificação (Tohge et al., 2013). A via do shikimate é altamente conservada sendo encontrada em fungos, bactérias e espécies vegetais onde opera na biossíntese não só dos três aminoácidos aromáticos descritos acima, mas também de inúmeros metabólitos secundários aromáticos como alcalóides, flavonóides, ligninas e antibióticos aromáticos. Muitos desses compostos são bioativos, além de desempenharem papéis importantes na defesa das plantas contra o estresse biótico e abiótico e as interações ambientais (Hamberger et al., 2006; Maeda e Dudareva, 2012), e como tal são altamente importantes do ponto de vista fisiológico. Estima-se que, em condições normais, até 20% do total de fluxos fixos de carbono fluem pela via do shikimate (Ni et al., 1996), com maior fluxo de carbono pela via em tempos de estresse vegetal ou crescimento rápido (Corea et al., 2012). Dada sua importância, talvez não seja surpreendente que todos os membros dos genes biossintéticos e enzimas correspondentes envolvidos na via do xiquimato tenham sido caracterizados em plantas modelo, como a Arabidopsis. A comparação de espécies cruzadas das enzimas biossintéticas do xiquimato revelou que estas partilham semelhanças de sequência, evolução divergente e uniformidade nos mecanismos de reacção (Dosselaere e Vanderleyden, 2001). No entanto, todas as outras espécies variam consideravelmente de fungos que desenvolveram um sistema complexo com um único polipeptídeo pentafuncional conhecido como complexo AroM que realiza cinco reacções consecutivas (Lumsden e Coggins, 1977; Duncan et al., 1987). Nesta revisão iremos resumir o conhecimento actual sobre a natureza genética desta via, focando as comparações entre espécies, fazendo a ponte entre uma vasta gama de espécies, incluindo algas (Chlamydomonas reinhardtii, Volvox carteri, Micromonas sp.., Ostreococcus tauri, Ostreococcus lucimarinus), musgo (Selaginella moellendorffii, Physcomitrella patens), monocotiledôneas (Sorghum bicolor, Zea mays, Brachypodium distachyon, Oryza sativa ssp. japonica e Oryza sativa ssp. indica), e dicots (Vitis vinifera, Theobroma cacao, Carica papaya, Arabidopsis thaliana, Arabidopsis lyrata, Populus trichocarpa, Ricinus communis, Manihot esculenta, Malus domestica, Fragaria vesca, Glycine max, Lotus japonicus, Medicago truncatula) (Tabela 1). Finalmente, comparamos e contrastamos a evolução desta via com a das vias mais especializadas da biossíntese fenilpropanoide.
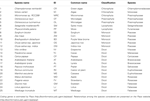
Tabela 1. Resumo das espécies utilizadas no estudo.
Shikimate Biosynthesis and Phenylalanine Derived Secondary Metabolism in Plants
Dado que os metabolitos fenólicos secundários que são derivados da fenilalanina via biossíntese do shikimate são amplamente distribuídos nas plantas e outras eucariotas, os genes que codificam as enzimas biossintéticas do shikimate são geralmente altamente conservados na natureza. Oito e duas reacções estão envolvidas na biossíntese do xiquimato e da fenilalanina, respectivamente. Ambos os membros de todas as famílias de genes e as correspondentes enzimas biossintéticas envolvidas nestas vias têm sido caracterizadas em plantas modelo como a Arabidopsis (Figura 1A). Em contraste, metabólitos fenólicos secundários derivados da fenilalanina apresentam uma distribuição considerável de espécies específicas com os metabólitos fenólicos secundários que têm sido encontrados no reino vegetal, tais como derivados da cumarina, monolignal, lignina, derivados da espermidina, flavonóide, estando o tanino presente em famílias específicas dentro da linhagem verde (Figura 1B). Esta diversidade tem surgido pela ação de diversas estratégias evolutivas, por exemplo, duplicação de genes e evolução cis-regulatória, a fim de se adaptar às condições ambientais predominantes. Dada a sua distribuição específica por espécie, os genes envolvidos no metabolismo secundário fenólico das plantas como a fenilamonia-lisase (PAL), a politase sintetase (PKS), as deoxigenases dependentes de 2-oxoglutarato (2ODD) e as UDP-glicosiltransferases (UGTs) são frequentemente utilizadas como estudos de caso de evolução vegetal (Tohge et al., 2013). Apesar de os genes biossintéticos shikimato-fenilalanina estarem bem conservados em todas as espécies, incluindo as algas, não foram detectados genes ortológicos fenólicos secundários relacionados com o metabolismo em todas as espécies de algas (Tabela 2, Tohge et al., 2013). Este resultado sugere uma origem consideravelmente mais antiga das vias xiquimato-fenilalanina. Nas próximas seções, discutiremos a evolução das vias do xiquimato-fenilalanina, focalizando a comparação entre espécies para cada gene codificado nas enzimas constituintes de cada via.
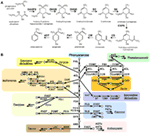
Figura 1. A biossíntese do metabolito secundário derivado do xiquimato e da fenilalanina nas plantas. (A) A biossíntese do xiquimato a partir do fosfoenolpiruvato (PEP) e D-erythrose 4-fosfato é descrita com genes caracterizados e metabólitos intermediários relatados. (B) biossíntese fenilalanina derivada da mebolita secundária fenólica maior na linhagem verde. A seta indica reação enzimática, o círculo indica metabólito. Abreviatura: DAHPS, 3-deoxi-D-arabino-heptulosonato 7-fosfato sintase; DQS, 3-de-hidroquinato sintase; DHQD/SD, 3-de-hidroquinato desidratase; SK, xiquimato quinase; ESPS, 3-fosfosiquimato 1-carboxiviniltransferase; CS, corismate sintetase; CM, corismate mutase; PAT, pré-fenato aminotransferase; ADT, arogenato desidratase. PAL, fenilalanina amonia-lisase; C4H, cana-4-hidroxilase; 4CL, 4-coumarato CoA ligase; CAD, cinamoyl-alcohol-desidrogenase; F5H, ferular 5-hidroxilase; C3H, cumarato 3-hidroxilase; ALDH, aldeído desidrogenase; CCR, cinamoyl-CoA redutase; HCT, hidroxicinamoyl-Coenzima A shikimato/quinato hidroxicinamoyltransferase; CCoAOMT, cafeoyl/CoA-3-O-metheltransferase; CHS, calcone-sintese; CHI, calcone-isomerase; F3H, flavanona 3-hidroxilase; F3′H, flavonoid-3′-hydroxylase; F3GT, flavonoid-3-O-glicosiltransferase; FS, flavone synthase; FOMT, flavonoid O-metiltransferase; FCGT, flavone-C-glycosyltransferase; FLS, flavonol synthese; F3GT, flavonoid-3-O-glycosyltransferase; DFR, diidroflavonol redutase; ANS, antocianidina sintetizada; AGT, flavonóide-O-glicosiltransferase; AAT, antocianina acyltransferase; BAN, oxidoreductase|dihydroflavonol reductase like; LAC, laccase.

Tabela 2. Genes biossintéticos e homólogos xiquimatos e fenilalanina em cada espécie com/sem genes duplicados em tandem.
3-Deoxi-D-Arabino-Heptulosonato 7-fosfato sintase
O primeiro passo enzimático da via do xiquimato, 3-deoxi-D-arabino-heptulosonato 7-fosfato sintase (DAHPS), catalisa uma condensação de aldol de fosfoenolpiruvato (PEP), e D-erythrose 4-fosfato (E4P) para produzir 3-deoxi-D-arabino-heptulosonato de 7-fosfato (DAHP) (Figura 1). De acordo com sua estrutura protéica, os DAHPS podem ser agrupados em duas classes distintas de homologia. Os DAHPSs da classe I derivados de micróbios contêm um corismate mutase (CM)-DAHPS bifuncional, por isso os DAHPSs microbianos, por exemplo, E. coli (AroF, G, e H) e S. cerevisiae (Aro3 e 4), são classificados como DAHPSs da classe I. Em contraste, DAHPSs classe II eram anteriormente considerados como presentes apenas em espécies vegetais, mas posteriormente foram relatados em certos micróbios como Streptomyces coelicolor, Streptomyces rimosus e Neurospora crassa (Bentley, 1990; Maeda e Dudareva, 2012). As actividades DAHPS (AroA) e CM (AroQ) de B. subtilis DAHPS estão, contudo, separadas por truncagem de domínio. Análise detalhada da estrutura sequencial das famílias de bactérias AroA e AroQ, estudos enzimáticos com a proteína completa e os domínios truncados de AroA e AroQ de B. subtilis, e comparação com as proteínas de fusão de Porphyromonas gingivalis, nas quais o domínio AroQ foi fundido ao terminal C de AroA, sugerem que a “regulação de feedback” pode ser de facto a ligação evolutiva entre as duas classes que evoluíram de membro primitivo não regulamentado da classe II DAHPS (Wu e Woodard, 2006). As DAHPSs de plantas de classe II têm sido relatadas a partir de raízes de cenoura (Suzich et al., 1985) e da cultura de células de batata (Pinto et al., 1986; Herrmann e Weaver, 1999). O DAHPS é codificado por três genes no genoma Arabidopsis (AtDAHPS1, AT4G39980; AtDAHPS2, At4g33510; AtDAHPS3, At1g22410). Consultas de busca de genes ortológicos usando os DAHPSs da Arabidopsis revelaram um único gene em espécies de algas (Chlamydomonas reinhardtii, Volvox carteri, Micromonas sp., e Ostreococcus tauri) e Lotus japonica, mas duas a oito isoformas em outras espécies de plantas superiores (Tabela 2). Os genes do tipo AtDAHPS1 e AtDAHPS2 apresentam expressão diferencial em Arabidopsis thaliana, Solanum lycopersicum, e Solanum tuberosum (Maeda e Dudareva, 2012). Os genes do tipo AtDAHPS1, que estão adicionalmente sujeitos à regulação redox pelo sistema de ferredoxina-teredoxina, exibem indução significativa por ferimento e infecção por patógenos (Keith et al., 1991; Gorlach et al., 1995; Maeda e Dudareva, 2012), enquanto os genes do tipo AtDAHPS2 exibem expressão constitutiva (Gorlach et al., 1995). Uma análise filogenética dos genes DAHPS revela quatro clades principais, (i) um clade de micrófitas, (ii) um clade de duplicação bryophyta, (iii) um clade de monocotiledôneas e dicotiledôneas, (iv) um clade de AtDAHPSs (Figura 2Aa). Além disso, o clade maior iv tem quatro subgrupos, (iv-a) grupo AtDAHPS2, (iv-b) monocot, (iv-c) grupo AtDAHPS1 e (iv-d) grupo AtDAHP3. Este resultado indica que os genes do tipo AtDAHPS1 expressos constitutivamente e os genes do tipo AtDAHPS 3 sensíveis ao estresse apresentam seqüência bem conservada entre as espécies (clade iv-c e iv-d), enquanto os genes do tipo AtDAHPS2 expressos constitutivamente estão claramente separados entre as espécies de monocotiledôneas e dicotiledôneas (clade iv-a).

Figura 2. Análise filogenética de árvores de shikimate e genes biossintéticos de fenilalanina em 25 espécies. Árvores filogenéticas da sequência de aminoácidos da via do xiquimato (A): (a), DAHPS, (b) DHS, (c) DHQD/SD, (d) SK, (e) ESPS, e (f) CS, (B) genes relacionados com a fenilalanina, (a) CM e (b) PAT. As sequências de aminoácidos dos genes biossintéticos do shikimate são obtidas a partir da base de dados Plaza (http://bioinformatics.psb.ugent.be/plaza/). As relações entre as espécies consideradas são apresentadas no site do Plaza. A árvore filogenética foi construída com as seqüências de proteínas alinhadas por MEGA (versão 5.10; http://www.megasoftware.net/; Kumar et al., 2004) usando o método de junção vizinha com os seguintes parâmetros: Correção de Poisson, deleção completa e bootstrap (1000 réplicas, semente aleatória). As seqüências de proteínas foram alinhadas pelo Plaza. Os valores nos ramos indicam suporte bootstrap em porcentagens.
3-Dehydroquinate Synthase
O segundo passo do caminho do shikimate é catalisado pela 3-dehydroquinate synthase (DHQS), uma enzima que promove a troca intramolecular do oxigênio do anel DAHP com carbono 7 para converter o DAHP em 3-dehydroquinate. Ao contrário da situação fúngica detalhada acima, o gene da planta DHQS é monofuncional e só é encontrado como uma única cópia em todas as espécies, com exceção da Glycine max que abriga dois genes em seu genoma (Figura 2Ab). A análise filogenética dos genes da DHQS revela três clades principais que consistem em (i) microfito (ii) briófito, (iii) monocotiledônea, (iv) Brassicaceae, e (v) espécies de dicotiledôneas. Intrigantemente, ao contrário de outros genes biossintéticos chiquimatos, a expressão gênica do gene DHQS não está bem correlacionada com a produção de fenilpropanoides em Arabidopsis (Hamberger et al., 2006).
3-Dehidroquinato Dehidratase/Shikimate Dehydrogenase
3-Deoxi-D-arabino-heptulosonato 7-fosfato é convertido em 3-deidroquinato pela enzima bifuncional 3-deidroquinato desidratase/hikimato desidrogenase (DHQD/SD), que catalisa em primeiro lugar a desidratação do DAHP a 3-hidroshikimato e consequentemente a redução reversível deste intermediário ahikimato utilizando o NADPH como co-factor. O DHQD/SD existe em três formas; shikimate desidrogenases classe I (tipo AroE), shikimate/quinate desidrogenases classe II (tipo YdiB) e classe III de shikimate desidrogenase-like (tipo SHD-L) (Michel et al., 2003; Singh et al., 2005). Em plantas classe IV, a atividade enzimática do DHQD é 10 vezes maior que a atividade do SD, indicando que a quantidade de 3-hidroxiquimato será mais que suficiente para suportar o fluxo através da via do xiquimato (Fiedler e Schultz, 1985). Esta enzima bifuncional desempenha um papel importante na regulação do metabolismo de várias vias fenólicas secundárias metabólicas (Bentley, 1990; Ding et al., 2007). Em geral, as plantas de semente contêm um único gene DHQD/SD que contém uma sequência que codifica um peptídeo de trânsito plástico em seu genoma (Maeda et al., 2011, Tabela 2). Entretanto, uma exceção a esta afirmação é Nicotiana tabacum, que contém dois genes em seu genoma. Intrigantemente, o silenciamento do NtDHD/SHD-1 resulta em forte inibição do crescimento e redução do nível de aminoácidos aromáticos, ácido clorogênico e conteúdo de lignina (Ding et al., 2007), entretanto, uma segunda isoforma citosólica pode compensar a produção de shikimate, mas não a nível fenotípico. Numa base mais geral, a análise filogenética revela que as microfitas também contêm um baixo número de genes DHQD/SD (entre um e dois), enquanto que uma separação clara entre (i) o clade da microfita, (ii) o clade da bryophyta, (iii) o clade da monocot, (iv) o clade da duplicação do gene tandem específico da espécie lenhosa, e (v) o clade da dicotiledônea pode ser observada (Figura 2Ac; Tabela 2). Curiosamente, a observação do clade de duplicação do gene tandem específico das espécies lenhosas sugere que estas espécies evoluíram após a duplicação do gene DHQD/SD. A localização citosólica do NtDHD/SHD-2 é intrigante já que a presença de DAHP synthase, ESPS synthase e isoformas CM sem seqüências de alvos de N-terminal plastidorisado tem sido relatada (d’Amato, 1984; Mousdale e Coggins, 1985; Ganson et al., 1986). Além disso, os achados de que tanto a ESPS synthase quanto a shikimate kinase (SK) estão ativas mesmo quando mantêm suas seqüências alvo (Dellacioppa et al., 1986; Schmid et al., 1992) sugerem que elas também poderiam ser potencialmente constituintes de uma via citosólica. Finalmente, experimentos nos quais mitocôndrias isoladas e altamente puras foram fornecidas com glicose 13C rotulada para investigar a ligação das isoformas citosólicas da glicólise (Giege et al., 2003) também revelou enriquecimento de 13C em shikimate (Sweetlove e Fernie, 2013), indicando que uma via citosólica completa também é provável nesta espécie.
Shikimate Kinase
A quinta reação da via do shikimate é catalisada pelo SK que catalisa a fosforilação dependente de ATP do shikimate para o shikimate 3-phospate (S3P). E. coli tem dois SKs, um de classe I (tipo AroL) e um de classe II (tipo AroK) que partilham apenas 30% da identidade da sequência (Griffin e Gasson, 1995; Whipp e Pittard, 1995; Herrmann e Weaver, 1999). Nas plantas, diferentes números de isoformas SK são encontrados em várias espécies; apenas uma em algas verdes, licófitas e briófitas, mas entre uma e três em plantas monocotiledóneas e dicotiledóneas (Tabela 2). Uma análise filogenética dos genes de SK apresenta cinco clades principais que consistem em (i) microfito, (ii) briófito, (iii) clade específico de espécies lenhosas de dicotiledôneas, (iv) clade de monocot e (v) clade de espécies de dicotiledôneas (Figura 2Ad). Anaylsis da proteína SK da Spinacia olerancea revelou que ela foi modulada pelo status energético e, portanto, é semelhante à proteína SK bacteriana e outras enzimas utilizadoras de ATP (Pacold e Anderson, 1973; Huang et al., 1975; Schmidt et al., 1990). Por esta razão, foi recentemente postulado que a SK pode estar ligada à energia que requer caminho de shikimate para o balanço energético celular (Maeda e Dudareva, 2012), no entanto, falta actualmente apoio experimental directo para esta hipótese. Em Arabidopsis, genes homólogos chamados SKL1 e SKL2, que são funcionalmente necessários para a biogênese cloroplástica, demonstraram ter surgido a partir da duplicação do gene SK (Fucile et al., 2008). Foram encontrados ortologs SKL1 e SKL2 em várias espécies de plantas-semente, mas não em algas verdes (Tabela 2).
5-enolypyruvylshikimate 3-fosfato Synthase
O 5-enolypyruvylshikimate 3-fosfato synthase (EPSPS, 3-fosfoshikimate 1-carboxi-vintltransferase) é o sexto passo e aqui uma segunda PEP é condensada com S3P para formar 5-enolpyruvylshiukimate 3-fosfato (EPSP). Como EPSPS é o único alvo conhecido para o herbicida glifosato (Steinrucken e Amrhein, 1980), isoformas desta enzima são frequentemente classificadas de acordo com sua sensibilidade ao glifosato, EPSPS sensível ao glifosato classe I está presente em bactérias e espécies vegetais, enquanto EPSPS insensível ao glifosato classe II que tem sido relatado em certas bactérias como Agrobacterium (Fucile et al., 2011). Nas plantas, um número diferente de isoformas EPSPS é encontrado em várias espécies; apenas uma única isoforma em algas verdes, licófitas e briófitas, mas uma ou duas são encontradas em espécies de monocotiledôneas e dicotiledôneas (Tabela 2). A análise filogenética dos genes EPSPS revelou, atipicamente para genes associados ao metabolismo do xiquimato, que cinco grandes grupos poderiam ser observados: (i) microfíte, (ii) bryophyte, (iii) clade específico de Brassicaceae, (iv) espécies de monocot e (v) clade de dicotiledôneas (Figura 2Ae). Há indicações claras de que genes EPSPS duplicados em Arabidopsis, maçã, videira, soja e álamo são o resultado de eventos de duplicação independentes dentro de suas linhagens, sendo ambas as cópias mantidas em Arabidopsis (Hamberger et al, 2006), entretanto, a razão para a divergência única neste gene da via é atualmente pouco clara.
Corismate Synthase
Chorismate, o produto final da via do shikimate, é subseqüentemente formado por corismate synthase (CS) que catalisa a eliminação do trans-1,4 fosfato do EPSP. Os CS são categorizados dentro de um de dois grupos funcionais (i) CS bifuncionais do tipo fúngico que estão associados à flavina-reductase dependente de NADPH ou (ii) CS monofuncionais do tipo bacteriano e vegetal (Schaller et al., 1991; Maeda e Dudareva, 2012). A reação catalisada por CS requer flavin mononucleotídeo (FMN) e sua reação geral é redox neutra (Ramjee et al., 1991; Macheroux et al., 1999; Maclean e Ali, 2003). O FMN representa um doador de elétrons para EPSP que facilita a clivagem do fosfato. O primeiro gene de CS da planta clonada foi o de C. sempervirens (Schaller et al., 1991) que contém um único CS em seu genoma. Dado que este gene tem uma sequência de sinais de importação de plastídeos 5′, estes resultados indicam que pode não haver um CS fora do plastídeo desta espécie. Pesquisando outras espécies revelou que um a dois genes de CS estavam presentes em algas verdes, licófitas e briófitas, bem como em espécies de dicotiledôneas, mas que um a três estão presentes nos genomas das espécies de maçã e leguminosas (Tabela 2). Uma análise filogenética dos genes de CS revela três grandes clades constituídos por (i) microfitas, (ii) monocotiledôneas, (iii) espécies de dicotiledôneas (Figura 2Af).
Corismate Mutase
Corismate Mutase catalisa o primeiro passo da biossíntese de fenilalanina e tirosina e, adicionalmente, representa um passo chave para a divisão do ramo da biossíntese de triptofano. CM catalisa a transformação do corismate em pré-fenisado através de um rearranjo Claisen. As proteínas CM menores bacterianas (tipo AroQ, CM classe I) apresentam atividade enzimática monofuncional, enquanto várias CM bifuncionais como CM-PDT, CM-PDH, e CM-DAHP foram adicionalmente encontradas em fungos e bactérias (CM classe II, Euverink et al., 1995; Romero et al., 1995; Chen et al., 2003; Baez-Viveros et al., 2004). Apesar de apenas um gene CM estar presente nos genomas das algas e licófitas, mais uma cópia de um único gene (dois a cinco) é encontrada em briófitas, bem como em espécies de monocotiledôneas e dicotiledôneas (Tabela 2). Nas plantas de semente, o CM1 tem um putativo peptídeo de trânsito plasmático, mas o CM2 não tem e é adicionalmente insensível à regulação alostérica por aminoácidos aromáticos (Benesova e Bode, 1992; Eberhard et al., 1996; Maeda e Dudareva, 2012). Várias espécies de plantas, especialmente as dicotiledôneas, têm um gene adicional da família CM3 que exibe uma alta semelhança de sequência com o CM2, mas carrega um suposto peptídeo de trânsito plasmático. Por exemplo, Arabidopsis tem três isozimas chamadas AtCM1 (At3g29200), AtCM2 (At5g10870), e AtCM3 (At1g69370) (Mobley et al., 1999; Tzin e Galili, 2010). A análise filogenética dos genes CS revela três clades principais que constituem (i) o clade AtCM2, (ii) o clade de micrófitas e briófitas, e (iii) o clade AtCM2 (Figura 2Ba). Além disso, o clade iii mostra dois subgrupos, (iii-a) subgrupos AtCM3 e (iii-b) subgrupo AtCM1 (Figura 2Ba) (Eberhard et al., 1996). Apesar disso, o subgrupo CM2 contém todas as espécies de plantas com sementes, as espécies de monocotiledôneas não estão contidas no subgrupo AtCM3. Recentemente a importância do CM foi estendida além do metabolismo intracelular, Em Zea mays, a corismate mutase Cmu1 secretada por Ustilago maydis, um patógeno amplamente caracterizado pelo desenvolvimento de grandes tumores de plantas e comumente conhecido como odor, é um fator de virulência. A absorção da proteína CMu1 do Ustilago pelas células vegetais permite o reencaminhamento do metabolismo vegetal e altera o estado metabólico destas células através do priming metabólico (Djamei et al., 2011). Agora parece que CMs secretadas são encontradas em muitos micróbios relacionados a plantas e esta forma de manipulação do hospedeiro parece ser uma arma geral no arsenal de patógenos vegetais.
Prefenato Aminotransferase e Arogenate Dehydratase
Prefenato aminotransferase (PAT) e a arogenate dehydratase (ADT) catalisam os passos finais para a produção de fenilalanina. Enquanto a ADT foi clonada pela primeira vez em 2007 (Cho et al., 2007; Huang et al., 2010), é apenas mais recentemente que a PAT foi clonada. Trabalhos publicados em 2011 identificaram a PAT em híbridos de Petunia, Arabidopsis thaliana e Solanum lycopersicum (Dal Cin et al., 2011; Maeda et al., 2011) e estabeleceram que ela direciona o fluxo de carbono do prefenato para o arogenado, mas também que é forte e coordenadamente upregulada com genes do metabolismo primário e voláteis do sabor derivado da fenilalanina. Em espécies vegetais, foi encontrado um número diferente de isoformas PAT. Embora as algas verdes contenham apenas um único gene PAT e ADT, as espécies monocotiledóneas têm entre um e dois PAT e entre dois e quatro ADT, enquanto os genomas das plantas dicotiledóneas contêm o mesmo número de PAT, mas dois a oito ADT (Tabela 2). A análise filogenética dos genes PAT mostra três grandes clades de (i) microfitas, (ii) monocotiledôneas, e (iii) espécies de dicotiledôneas (Figura 2Bb).
Genes envolvidos em metabolismos secundários fenólicos vegetais
Babolismo secundário fenólico exibe uma imensa diversidade química devido à evolução dos genes enzimáticos que estão envolvidos nas várias vias biossintéticas e decorativas. Tal variação é causada pela diversidade e redundância de vários genes chave do metabolismo secundário fenólico, tais como PKSs, citocromo P450s (CYPs), dioxigenases dependentes de Fe2+/2-oxoglutarato (2ODDs), e UDP-glicosiltransferases (UGTs). Por outro lado, existem outros genes biossintéticos gerais relacionados com fenilpropanoides, fenilalanina amonia-lisase (PAL), cinamato 4-hidroxilase (C4H), e 4-coumarato:coenzima A ligase (4CL), que são necessários para diferenciar várias classes de metabolismo secundário fenólico. Todos esses genes centrais codificam enzimas importantes que ativam um número de ácidos hidroxicinâmicos para fornecer precursores para a biossíntese de ligninas, monolinais e, de fato, todos os outros principais metabolitos fenólicos secundários em plantas superiores (Lozoya et al., 1988; Allina et al., 1998; Hu et al., 1998; Ehlting et al., 1999; Lindermayr et al., 2002; Hamberger e Hahlbrock, 2004). Dado que o metabolismo fenólico secundário apresenta uma especificidade considerável das espécies, a investigação dos genes que codificam as enzimas biossintéticas responsáveis é frequentemente utilizada como exemplo de quimiotaxonomia para a compreensão da evolução das plantas. No entanto, considerando a evolução destes genes isoladamente é bastante restritiva uma compreensão mais profunda, combinando isto com a investigação da evolução dos genes biossintéticos xiquimato-fenilalanina na linhagem verde.
Conclusão
Durante o longo período evolutivo coberto de algas aquáticas a plantas terrestres, as plantas adaptaram-se aos nichos ambientais com as estratégias evolutivas como a duplicação de genes e a evolução convergente pela filtração da selecção natural. Os genes da biossíntese do shikimate das plantas evoluíram em conformidade (Figura 3). Nesta revisão, demonstramos que os genes biossintéticos do metabolismo primário dos aminoácidos aromáticos estão bem conservados entre as algas e todas as plantas terrestres. Entretanto, ao contrário das espécies de algas que não possuem isoformas nem genes duplicados em seus genomas, todas as plantas terrestres abrigam duplicações de genes incluindo duplicações de genes tandem que são particularmente proeminentes nos casos de DAHPS, DHQD/SD, CS, CM, e ADT (Figura 3A; Tabela 2). Nossa análise filogenética revelou uma clara separação entre algas, monocotiledôneas, dicotiledôneas, espécies lenhosas e plantas leguminosas. A análise da presença e do número de cópias de genes-chave através destas espécies dá várias dicas de como melhorar a nossa compreensão do andaime a partir do qual estes genes evoluíram. No entanto, as pressões evolutivas exatas sobre os genes da biossíntese do xiquimato, incluindo a ocorrência única do complexo Arom, exigirão estudos adicionais consideráveis. Dito isto é intrigante comparar e contrastar os genes biossintéticos dos que se encontram a jusante deles na produção de fenólicos vegetais (Figura 3B). Curiosamente, os genes da via shikimate são onipresentes através da linhagem verde, enquanto isto não pode ser dito para todos os genes a jusante da biossíntese fenilpropanoide. Além disso, há uma duplicação muito maior de genes dentro da biossíntese fenilpropanoide do que a biossíntese do xiquimato (Figura 3A; Tabela 2). Este fato também se reflete no nível de diversidade química das respectivas vias com a essencialidade da via do xiquimato impedindo muita diversidade, mas as espécies fenilpropanoides muitas vezes são redundantes em função umas das outras. Parece provável que a via fenilpropanoide tenha surgido inicialmente através de mutações que se acumularam nos genes da via do xiquimato. Entretanto, enquanto estes foram potencialmente benéficos em plantas terrestres por razões que discutimos em nossa recente revisão destes compostos (Tohge et al., 2013) eles não parecem compartilhar a essencialidade do xiquimato em toda a linhagem verde.
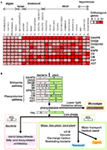
Figure 3. Mapa de calor para isoformas de genes biossintéticos de shikimato-fenilalanina em genomas vegetais e esquema hipotético para a evolução do metabolismo fenólico secundário derivado da fenilalanina. (A) Mapa do número de isoformas do gene biossintético da shikimato-fenilalanina em 25 espécies. (B) Figura esquemática hipotética dos genes biossintéticos de xiquimato-fenilalanina e a sua evolução do metabolismo secundário fenólico.
Conflito de interesses
Os autores declaram que a pesquisa foi realizada na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
Conhecimento
Atividade de pesquisa de Takayuki Tohge é apoiada pela Fundação Alexander von Humboldt. O financiamento da Max-Planck-Society (para Takayuki Tohge, Mutsumi Watanabe, Rainer Hoefgen, Alisdair R. Fernie) é reconhecido com gratidão.
Benesova, M., and Bode, R. (1992). Chorismate mutase isoforms from seeds and seedlings of Papaver somniferum. Phytochemistry 31, 2983-2987.
CrossRef Full Text
Fiedler, E., e Schultz, G. (1985). Localização, purificação e caracterização do shikimate oxidoreductase-dehydroquinate hidrolase a partir de estroma de espinafrocloroplastos. Plant Physiol. 79, 212-218.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Hamberger, B., Ehlting, J., Barbazuk, B., e Douglas, C. J. (2006). Comparative genomics of the shikimate pathway in Arabidopsis, Populus trichocarpa and Oryza sativa: shikimate pathway gene family structure and identification of candidates for missing links in phenylalanine biosynthesis. Adv. recente. Fitoquímica. 40, 85-113.
CrossRef Full Text
Maclean, J., e Ali, S. (2003). A estrutura do corismate synthase revela um novo flavin binding a uma reação química única. Estrutura 11, 1499-1511.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Schmid, J., Schaller, A., Leibinger, U., Boll, W., and Amrhein, N. (1992). O precursor do shikimato-cinase do tomate sintetizado in vitro é enzimaticamente ativo e é importado e processado para a enzima madura por cloroplastos. Planta J. 2, 375-383.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Tzin, V., e Galili, G. (2010). Novos insights sobre os caminhos da biossíntese dos xiquimatos e aminoácidos aromáticos nas plantas. Mol. Planta 3, 956-972.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text