Introduzione
La via dello shikimate è strettamente interconnessa con quelle degli aminoacidi aromatici (L-triptofano, L-fenilalanina e L-tirosina) e nelle piante terrestri sopporta flussi molto elevati con stime della quantità di carbonio fisso che passa attraverso la via che varia tra il 20 e il 50% (Weiss, 1986; Corea et al., 2012; Maeda e Dudareva, 2012). Una notevole attenzione della ricerca è stata posta su questa via poiché gli aminoacidi aromatici non sono prodotti dall’uomo e dal bestiame monogastrico e sono quindi un importante componente della dieta (Tzin e Galili, 2010). Inoltre, uno degli enzimi del percorso – 5-enolpyruvalshikimate-3-fosfato sintasi (EPSP) – è uno dei siti bersaglio erbicida più ampiamente utilizzato (vedi, Duke e Powles, 2008). Inoltre, come abbiamo recentemente descritto, i metaboliti secondari fenolici delle piante e i loro precursori sono sintetizzati attraverso la via della biosintesi shikimate e i suoi numerosi punti di diramazione (Tohge et al., 2013). La via dello shikimate è altamente conservata e si trova in funghi, batteri e specie di piante dove opera nella biosintesi non solo dei tre aminoacidi aromatici descritti sopra ma anche di innumerevoli metaboliti secondari aromatici come alcaloidi, flavonoidi, lignine e antibiotici aromatici. Molti di questi composti sono bioattivi e svolgono ruoli importanti nella difesa delle piante contro gli stress biotici e abiotici e le interazioni ambientali (Hamberger et al., 2006; Maeda e Dudareva, 2012), e come tali sono altamente fisiologici. Si stima che in condizioni normali fino al 20% del carbonio totale fissato passi attraverso la via dello shikimate (Ni et al., 1996), con un maggiore flusso di carbonio attraverso la via in periodi di stress della pianta o di rapida crescita (Corea et al., 2012). Data la sua importanza, forse non è sorprendente che tutti i membri dei geni biosintetici e i corrispondenti enzimi coinvolti nella via shikimate siano stati caratterizzati in piante modello come l’Arabidopsis. Il confronto interspecie degli enzimi biosintetici shikimate ha rivelato che essi condividono la somiglianza di sequenza, l’evoluzione divergente e la comunanza nei meccanismi di reazione (Dosselaere e Vanderleyden, 2001). Tuttavia, tutte le altre specie variano considerevolmente dai funghi che hanno evoluto un sistema complesso con un singolo polipeptide pentafunzionale noto come il complesso AroM che esegue cinque reazioni consecutive (Lumsden e Coggins, 1977; Duncan et al., 1987). In questa revisione riassumeremo le conoscenze attuali riguardanti la natura genetica di questo percorso concentrandoci su confronti tra specie diverse, comprese le alghe (Chlamydomonas reinhardtii, Volvox carteri, Micromonas sp, Ostreococcus tauri, Ostreococcus lucimarinus), muschi (Selaginella moellendorffii, Physcomitrella patens), monocotiledoni (Sorghum bicolor, Zea mays, Brachypodium distachyon, Oryza sativa ssp. japonica e Oryza sativa ssp. indica), e dicotiledoni (Vitis vinifera, Theobroma cacao, Carica papaya, Arabidopsis thaliana, Arabidopsis lyrata, Populus trichocarpa, Ricinus communis, Manihot esculenta, Malus domestica, Fragaria vesca, Glycine max, Lotus japonicus, Medicago truncatula) (Tabella 1). Infine, confrontiamo e contrastiamo l’evoluzione di questa via con quella delle vie più specializzate della biosintesi dei fenilpropanoidi.
Tabella 1. Riassunto delle specie usate nello studio.
Biosintesi shikimate e metabolismo secondario derivato dalla fenilalanina nelle piante
Dato che i metaboliti secondari fenolici che sono derivati dalla fenilalanina attraverso la biosintesi shikimate sono ampiamente distribuiti nelle piante e altri eucarioti, i geni che codificano gli enzimi biosintetici shikimate sono generalmente altamente conservati in natura. Otto e due reazioni sono coinvolte nella biosintesi di shikimate e fenilalanina, rispettivamente. Sia i membri di tutte le famiglie di geni che i corrispondenti enzimi biosintetici coinvolti in queste vie sono stati caratterizzati in piante modello come l’Arabidopsis (Figura 1A). Al contrario, i metaboliti secondari fenolici derivati dalla fenilalanina mostrano una notevole distribuzione specie-specifica con i metaboliti secondari fenolici sono stati trovati nel regno vegetale come i derivati della cumarina, monolignale, lignina, derivati della spermidina, flavonoide, tannino essendo presenti in famiglie specifiche all’interno della stirpe verde (Figura 1B). Questa diversità è nata dall’azione di diverse strategie evolutive, per esempio la duplicazione dei geni e l’evoluzione cis-regolatoria per adattarsi alle condizioni ambientali prevalenti. Data la loro distribuzione specie-specifica, i geni coinvolti nel metabolismo secondario fenolico delle piante come la fenilammonia-liasi (PAL), la sintasi dei policheti (PKS), le deossigenasi dipendenti dal 2-ossoglutarato (2ODD) e le UDP-glicosiltransferasi (UGT) sono spesso utilizzati come casi di studio dell’evoluzione delle piante (Tohge et al., 2013). Nonostante il fatto che i geni biosintetici della shikimate-fenilalanina siano ben conservati in tutte le specie, comprese quelle algali, i geni ortologhi legati al metabolismo secondario fenolico non sono stati rilevati in tutte le specie algali (Tabella 2, Tohge et al., 2013). Questo risultato suggerisce un’origine molto più antica delle vie shikimate-fenilalanina. Nelle prossime sezioni, discuteremo l’evoluzione delle vie shikimate-fenilalanina concentrandoci sui confronti tra specie diverse per ogni gene che codifica uno degli enzimi costitutivi di entrambe le vie.
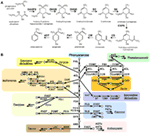
Figura 1. La biosintesi dei metaboliti secondari derivati da shikimate e fenilalanina nelle piante. (A) La biosintesi di shikimate a partire da fosfoenolpiruvato (PEP) e D-eritrosio 4-fosfato è descritta con geni caratterizzati e metaboliti intermedi riportati. (B) biosintesi del mebolita secondario fenilalanina derivato principale fenolico nel lignaggio verde. La freccia indica la reazione enzimatica, il cerchio indica il metabolita. Abbreviazione: DAHPS, 3-deossi-D-arabino-eptulosonato 7-fosfato sintasi; DQS, 3-deidroquinato sintasi; DHQD/SD, 3-deidroquinato deidratasi; SK, shikimate chinasi; ESPS, 3-fosfoschimato 1-carbossiviniltransferasi; CS, corismato sintasi; CM, corismato mutasi; PAT, prefenato aminotransferasi; ADT, arogenato deidratasi. PAL, fenilalanina ammoniaca-liasi; C4H, cinnamato-4-idrossilasi; 4CL, 4-cumarato CoA ligasi; CAD, cinnamoil-alcol deidrogenasi; F5H, ferulato 5-idrossilasi; C3H, cumarato 3-idrossilasi; ALDH, aldeide deidrogenasi; CCR, cinnamoil-CoA reduttasi; HCT, idrossicinnamoil-Coenzima A shikimate/quinato idrossicinnamoiltransferasi; CCoAOMT, caffeoil/CoA-3-O-metiltransferasi; CHS, chalcone synthese; CHI, chalcone isomerase; F3H, flavanone 3-idrossilasi; F3′H, flavonoide-3′-idrossilasi; F3GT, flavonoide-3-O-glicosiltransferasi; FS, flavone sintasi; FOMT, flavonoide O-metiltransferasi; FCGT, flavone-C-glicosiltransferasi; FLS, flavonolo synthese; F3GT, flavonoide-3-O-glicosiltransferasi; DFR, diidroflavonolo reduttasi; ANS, Anthocyanidin synthese; AGT, flavonoide-O-glicosiltransferasi; AAT, antocianina aciltransferasi; BAN, ossidoreduttasi|diidroflavonolo reduttasi come; LAC, laccasi.

Tabella 2. Geni biosintetici di shikimate e fenilalanina e omologhi in ogni specie con/senza geni duplicati in tandem.
3-Deossi-D-Arabino-Eptulosonato 7-fosfato sintasi
Il primo passo enzimatico della via dello shikimate, 3-deossi-D-arabino-epulosonato 7-fosfato sintasi (DAHPS), catalizza una condensazione aldolica del fosfoenolpiruvato (PEP) e del D-eritrosio 4-fosfato (E4P) per produrre 3-deossi-D-arabino-eptulosonato 7-fosfato (DAHP) (Figura 1). Secondo la loro struttura proteica, i DAHPS possono essere raggruppati in due classi omologiche distinte. I DAHPS di classe I derivati dai microbi contengono un dominio bifunzionale chorismate mutase (CM)-DAHPS, per questo motivo i DAHPS microbici, per esempio, E. coli (AroF, G, e H) e S. cerevisiae (Aro3 e 4), sono classificati come DAHPS di classe I. Al contrario, i DAHPS di classe II erano precedentemente ritenuti presenti solo nelle specie vegetali, ma sono stati successivamente riportati in alcuni microbi come Streptomyces coelicolor, Streptomyces rimosus, e Neurospora crassa (Bentley, 1990; Maeda e Dudareva, 2012). Le attività DAHPS (AroA) e CM (AroQ) di B. subtilis DAHPS sono, tuttavia, separate dal troncamento del dominio. L’analisi dettagliata della struttura della sequenza delle famiglie batteriche AroA e AroQ, studi enzimatici con la proteina completa e i domini troncati di AroA e AroQ di B. subtilis, e il confronto con proteine di fusione di Porphyromonas gingivalis in cui il dominio AroQ è stato fuso al terminale C di AroA, suggeriscono che la “regolazione del feedback” può effettivamente essere il legame evolutivo tra le due classi che si sono evolute dal membro primitivo non regolato della classe II DAHPS (Wu e Woodard, 2006). I DAHPS delle piante di classe II sono stati riportati dalle radici delle carote (Suzich et al., 1985) e dalle colture cellulari delle patate (Pinto et al., 1986; Herrmann e Weaver, 1999). DAHPS è codificato da tre geni nel genoma di Arabidopsis (AtDAHPS1, AT4G39980; AtDAHPS2, At4g33510; AtDAHPS3, At1g22410). Le query di ricerca dei geni omologhi utilizzando i DAHPS dell’Arabidopsis, hanno rivelato un singolo gene nelle specie di alghe (Chlamydomonas reinhardtii, Volvox carteri, Micromonas sp., e Ostreococcus tauri) e Lotus japonica ma da due a otto isoforme in altre specie di piante superiori (Tabella 2). I geni di tipo AtDAHPS1 e AtDAHPS2 mostrano un’espressione differenziale in Arabidopsis thaliana, Solanum lycopersicum e Solanum tuberosum (Maeda e Dudareva, 2012). I geni di tipo AtDAHPS1, che sono inoltre soggetti alla regolazione redox da parte del sistema ferredoxina-tiorodossina, mostrano una significativa induzione da ferite e infezioni da patogeni (Keith et al., 1991; Gorlach et al., 1995; Maeda e Dudareva, 2012), mentre i geni di tipo AtDAHPS2 mostrano un’espressione costitutiva (Gorlach et al., 1995). Un’analisi filogenetica dei geni DAHPS rivela quattro cladi principali, (i) un clade microfita, (ii) un clade briofita duplicazione, (iii) monocot e dicot specie legnose clade, (iv) un clade AtDAHPSs (Figura 2Aa). Inoltre, il clade principale iv ha quattro sottogruppi, (iv-a) gruppo AtDAHPS2, (iv-b) monocot, (iv-c) gruppo AtDAHPS1 e (iv-d) gruppo AtDAHP3. Questo risultato indica che i geni costitutivamente espressi AtDAHPS1 e il tipo AtDAHPS 3 sensibile allo stress mostrano una sequenza ben conservata tra le specie (clade iv-c e iv-d), mentre i secondi geni costitutivamente espressi AtDAHPS2 sono chiaramente separati tra le specie monocot e dicot (clade iv-a).

Figura 2. Analisi dell’albero filogenetico dei geni biosintetici di shikimate e fenilalanina in 25 specie. Alberi filogenetici delle sequenze di aminoacidi di (A) shikimate pathway: (a), DAHPS, (b) DHS, (c) DHQD/SD, (d) SK, (e) ESPS, e (f) CS, (B) geni correlati alla fenilalanina, (a) CM e (b) PAT. Le sequenze di amminoacidi dei geni biosintetici shikimate sono ottenute dal database Plaza (http://bioinformatics.psb.ugent.be/plaza/). Le relazioni tra le specie considerate sono presentate sul sito web Plaza. L’albero filogenetico è stato costruito con le sequenze proteiche allineate da MEGA (versione 5.10; http://www.megasoftware.net/; Kumar et al., 2004) utilizzando il metodo neighbor-joining con i seguenti parametri: Correzione di Poisson, cancellazione completa, e bootstrap (1000 repliche, seme casuale). Le sequenze proteiche sono state allineate da Plaza. I valori sui rami indicano il supporto bootstrap in percentuale.
3-Deidroquinato sintasi
La seconda fase della via shikimate è catalizzata dalla 3-deidroquinato sintasi (DHQS), un enzima che promuove lo scambio intramolecolare dell’ossigeno dell’anello DAHP con il carbonio 7 per convertire DAHP in 3-deidroquinato. A differenza della situazione fungina descritta sopra, il gene DHQS delle piante è monofunzionale e si trova solo in una singola copia in tutte le specie ad eccezione di Glycine max che ospita due geni nel suo genoma (Figura 2Ab). L’analisi filogenetica dei geni DHQS rivela tre cladi principali costituiti da (i) microfite (ii) briofite, (iii) monocot, (iv) Brassicaceae e (v) specie dicot. Curiosamente, al contrario di altri geni biosintetici shikimate, l’espressione genica del gene DHQS non è ben correlata alla produzione di fenilpropanoidi in Arabidopsis (Hamberger et al., 2006).
3-Deidroquinate Dehydratase/Shikimate Dehydrogenase
3-Deoxy-D-arabino-heptulosonate 7-phosphate è convertito in 3-deidroquinate dall’enzima bifunzionale 3-dehydroquinate dehydratase/shikimate dehydrogenase (DHQD/SD), che catalizza in primo luogo la disidratazione di DAHP a 3-deidroshikimate e di conseguenza la riduzione reversibile di questo intermedio a shikimate usando NADPH come cofattore. DHQD/SD esiste in tre forme; shikimate deidrogenasi di classe I specifica per i batteri (tipo AroE), shikimate/quinate deidrogenasi di classe II (tipo YdiB), e shikimate dehydrogenase-like di classe III (tipo SHD-L) (Michel et al., 2003; Singh et al., 2005). Nelle piante di classe IV, l’attività enzimatica della DHQD è 10 volte superiore all’attività della SD, indicando che la quantità di 3-deidroshikimate sarà più che sufficiente per sostenere il flusso attraverso la via dello shikimate (Fiedler e Schultz, 1985). Questo enzima bifunzionale gioca un ruolo importante nella regolazione del metabolismo di diverse vie metaboliche secondarie fenoliche (Bentley, 1990; Ding et al., 2007). In generale, le piante da seme contengono un singolo gene DHQD/SD che contiene una sequenza che codifica un peptide di transito plastico nel loro genoma (Maeda et al., 2011, Tabella 2). Tuttavia, un’eccezione a questa affermazione è Nicotiana tabacum che contiene due geni nel suo genoma. Intrigante, il silenziamento di NtDHD/SHD-1 risulta una forte inibizione della crescita e una riduzione del livello di aminoacidi aromatici, acido clorogenico e contenuto di lignina (Ding et al., 2007), tuttavia, una seconda isoforma citosolica può compensare la produzione di shikimate ma non a livello fenotipico. Su una base più generale l’analisi filogenetica rivela che le microfite contengono anche un basso numero di geni DHQD/SD (tra uno e due), mentre una chiara separazione tra (i) il clade delle microfite, (ii) il clade delle briofite, (iii) il clade delle monocot, (iv) il clade delle specie legnose con duplicazione genica in tandem, e (v) il clade delle dicot (Figura 2Ac; Tabella 2). È interessante notare che l’osservazione del clade di duplicazione dei geni in tandem specifico delle specie legnose suggerisce che queste specie si sono evolute dopo la duplicazione del gene DHQD/SD. La localizzazione citosolica di NtDHD/SHD-2 è intrigante poiché è stata riportata la presenza di DAHP sintasi, ESPS sintasi e CM isoforme prive di sequenze di targeting plastidico N-terminali (d’Amato, 1984; Mousdale e Coggins, 1985; Ganson et al., 1986). Inoltre, i risultati che sia l’ESPS sintasi che la shikimate chinasi (SK) sono attive anche quando mantengono le loro sequenze bersaglio (Dellacioppa et al., 1986; Schmid et al., 1992) suggeriscono che potrebbero anche essere potenzialmente costituenti di un percorso citosolico. Infine, gli esperimenti in cui i mitocondri isolati e altamente puri sono stati forniti di glucosio marcato con 13C per studiare il legame delle isoforme citosoliche della glicolisi (Giege et al, 2003) ha anche rivelato un arricchimento di 13C in shikimate (Sweetlove e Fernie, 2013), indicando che un percorso citosolico completo è probabile anche in questa specie.
Shikimate Kinase
La quinta reazione del percorso shikimate è catalizzata da SK che catalizza la fosforilazione ATP-dipendente di shikimate a shikimate 3-fosfato (S3P). E. coli ha due SK, uno di classe I (tipo AroL) e uno di II (tipo AroK) che condividono solo il 30% di identità di sequenza (Griffin e Gasson, 1995; Whipp e Pittard, 1995; Herrmann e Weaver, 1999). Nelle piante, un numero diverso di isoforme SK si trova in diverse specie; solo una nelle alghe verdi, nelle licofite e nelle briofite ma tra una e tre nelle piante monocotiledoni e dicotiledoni (Tabella 2). Un’analisi filogenetica dei geni SK presenta cinque cladi principali costituiti da (i) microfite, (ii) briofite, (iii) clade di specie legnose dicot, (iv) clade monocot e (v) clade di specie dicot (Figura 2Ad). L’analisi della proteina SK di Spinacia olerancea ha rivelato che è modulata dallo stato energetico ed è quindi simile alla proteina SK batterica e ad altri enzimi che utilizzano ATP (Pacold e Anderson, 1973; Huang et al., 1975; Schmidt et al., 1990). Per questo motivo è stato recentemente postulato che SK può collegare la via shikimate che richiede energia al bilancio energetico cellulare (Maeda e Dudareva, 2012), tuttavia, il supporto sperimentale diretto per questa ipotesi è attualmente mancante. In Arabidopsis, i geni omologhi chiamati SKL1 e SKL2, che sono funzionalmente richiesti per la biogenesi dei cloroplasti, hanno dimostrato di essere sorti dalla duplicazione del gene SK (Fucile et al., 2008). Gli ortologhi SKL1 e SKL2 sono stati trovati in diverse specie di piante da seme, ma non nelle alghe verdi (Tabella 2).
5-enolypyruvylshikimate 3-Phosphate Synthase
La 5-enolypyruvylshikimate 3-phosphate synthase (EPSPS, 3-phosphoshikimate 1-carboxyvintltransferase) è il sesto passo e qui una seconda PEP viene condensata con S3P per formare 5-enolpyruvylshiukimate 3-phosphate (EPSP). Poiché EPSPS è l’unico bersaglio noto per l’erbicida glifosato (Steinrucken e Amrhein, 1980), le isoforme di questo enzima sono spesso classificate in base alla loro sensibilità al glifosato, EPSPS sensibile al glifosato di classe I è presente in batteri e specie vegetali, mentre EPSPS insensibile al glifosato di classe II che è stato riportato in alcuni batteri come Agrobacterium (Fucile et al., 2011). Nelle piante, un numero diverso di isoforme EPSPS si trova in diverse specie; solo una singola isoforma nelle alghe verdi, nelle licofite e nelle briofite, ma una o due si trovano nelle specie monocot e dicot (Tabella 2). L’analisi filogenetica dei geni EPSPS ha rivelato, atipicamente per i geni associati al metabolismo shikimate, che cinque gruppi principali potrebbero essere osservati; (i) microfita, (ii) briofita, (iii) clade specifico Brassicaceae, (iv) specie monocot, e (v) clade specie dicot (Figura 2Ae). Ci sono chiare indicazioni che i geni EPSPS duplicati in Arabidopsis, melo, vite, soia e pioppo sono il risultato di eventi di duplicazione indipendenti all’interno dei loro lignaggi con entrambe le copie mantenute in Arabidopsis (Hamberger et al., 2006), tuttavia, la ragione della divergenza unica in questo gene del percorso non è attualmente chiara.
Corismato sintasi
Corismato, il prodotto finale del percorso shikimate, è successivamente formato dalla chorismato sintasi (CS) che catalizza l’eliminazione trans-1,4 del fosfato dall’EPSP. I CS sono classificati all’interno di uno dei due gruppi funzionali (i) CS bifunzionali di tipo fungino che sono associati con la flavin reduttasi NADPH-dipendente o (ii) CS monofunzionali di tipo batterico e vegetale (Schaller et al., 1991; Maeda e Dudareva, 2012). La reazione catalizzata dal CS richiede il mononucleotide di flavina (FMN) e la sua reazione complessiva è redox neutra (Ramjee et al., 1991; Macheroux et al., 1999; Maclean e Ali, 2003). Il FMN rappresenta un donatore di elettroni per l’EPSP che facilita la scissione del fosfato. Il primo gene CS vegetale clonato è stato quello di C. sempervirens (Schaller et al., 1991) che contiene un solo CS nel suo genoma. Dato che questo gene ha una sequenza di segnale di importazione del plastide al 5′, questi risultati indicano che non ci può essere CS al di fuori del plastide di questa specie. L’indagine su altre specie ha rivelato che da uno a due geni CS sono presenti nelle alghe verdi, nelle licofite e nelle briofite così come nelle specie dicot, ma che da uno a tre sono presenti nei genomi delle specie di mele e leguminose (Tabella 2). Un’analisi filogenetica dei geni CS rivela tre cladi principali costituiti da (i) microfite, (ii) monocot, (iii) specie dicot (Figura 2Af).
Corismato mutasi
Corismato mutasi catalizza il primo passo della biosintesi di fenilalanina e tirosina e rappresenta inoltre un passo chiave verso la divisione del ramo della biosintesi del triptofano. Il CM catalizza la trasformazione del corismato in prefenato attraverso un riarrangiamento di Claisen. Le proteine CM minori batteriche (tipo AroQ, classe I CM) mostrano un’attività enzimatica monofunzionale, mentre diverse CM bifunzionali come CM-PDT, CM-PDH e CM-DAHP sono state trovate anche in funghi e batteri (classe II CM, Euverink et al., 1995; Romero et al., 1995; Chen et al., 2003; Baez-Viveros et al., 2004). Nonostante il fatto che un solo gene CM sia presente nei genomi delle alghe e delle licofite, più copie di un solo gene (da due a cinque) si trovano nelle briofite e nelle specie monocotiledoni e dicotiledoni (tabella 2). Nelle piante da seme, il CM1 porta un peptide putativo di transito plastidico, ma il CM2 non lo fa ed è inoltre solitamente insensibile alla regolazione allosterica da parte degli aminoacidi aromatici (Benesova e Bode, 1992; Eberhard et al., 1996; Maeda e Dudareva, 2012). Diverse specie di piante, in particolare le piante dicot, hanno un gene aggiuntivo della famiglia CM3 che mostra un’elevata somiglianza di sequenza con CM2, ma porta un peptide putativo di transito plastidico. Per esempio, l’Arabidopsis ha tre isozimi chiamati AtCM1 (At3g29200), AtCM2 (At5g10870) e AtCM3 (At1g69370) (Mobley et al., 1999; Tzin e Galili, 2010). Analisi filogenetica dei geni CS rivela tre cladi principali che costituiscono (i) clade AtCM2, (ii) clade microfite e briofite e (iii) clade AtCM2 (Figura 2Ba). Inoltre, il clade iii mostra due sottogruppi, (iii-a) sottogruppo AtCM3 e (iii-b) sottogruppo AtCM1 (Figura 2Ba) (Eberhard et al., 1996). Nonostante il sottogruppo CM2 contenga tutte le specie di piante da seme, le specie monocotiledoni non sono contenute nel sottogruppo AtCM3. Recentemente l’importanza del CM è stata estesa oltre il metabolismo intracellulare, In Zea mays, la chorismate mutase Cmu1 secreta da Ustilago maydis, un patogeno diffuso caratterizzato dallo sviluppo di grandi tumori vegetali e comunemente noto come smut, è un fattore di virulenza. L’assorbimento della proteina Ustilago CMu1 da parte delle cellule delle piante permette di reindirizzare il metabolismo delle piante e cambia lo stato metabolico di queste cellule attraverso un priming metabolico (Djamei et al., 2011). Sembra ora che i CM secreti si trovino in molti microbi legati alle piante e questa forma di manipolazione dell’ospite sembrerebbe essere un’arma generale nell’arsenale dei patogeni delle piante.
Prefenato Aminotransferasi e Arogenato Deidratasi
Prefenato aminotransferasi (PAT) e arogenato deidratasi (ADT) catalizzano le fasi finali della produzione di fenilalanina. Mentre ADT è stata clonata per la prima volta nel 2007 (Cho et al., 2007; Huang et al., 2010), è solo più recentemente che PAT è stata clonata. Documenti pubblicati nel 2011 hanno identificato PAT in Petunia hybrid, Arabidopsis thaliana e Solanum lycopersicum (Dal Cin et al., 2011; Maeda et al., 2011) e hanno stabilito che dirige il flusso di carbonio dal prefenato all’arogenato, ma anche che è fortemente e coordinatamente upregolato con i geni del metabolismo primario e i volatili da sapore derivati dalla fenilalanina. Nelle specie vegetali, è stato trovato un diverso numero di isoforme PAT. Anche se le alghe verdi contengono solo singoli geni PAT e ADT, le specie monocotiledoni hanno da uno a due PAT e da due a quattro ADT, mentre i genomi delle piante dicotiledoni contengono lo stesso numero di PAT ma da due a otto ADT (tabella 2). L’analisi filogenetica dei geni PAT mostra tre cladi principali di (i) microfite, (ii) monocot, e (iii) specie dicot (Figura 2Bb).
Geni coinvolti nei metabolismi secondari fenolici delle piante
Il metabolismo secondario fenolico mostra una immensa diversità chimica dovuta all’evoluzione dei geni enzimatici che sono coinvolti nelle varie vie biosintetiche e decorative. Tale variazione è causata dalla diversità e dalla ridondanza di diversi geni chiave del metabolismo secondario fenolico come le PKS, i citocromi P450 (CYP), le diossigenasi dipendenti da Fe2+/2-oxoglutarate (2ODD) e le UDP-glicosiltransferasi (UGT). D’altra parte, ci sono altri geni biosintetici generali legati ai fenilpropanoidi, fenilalanina ammoniaca-liasi (PAL), cinnamato 4-idrossilasi (C4H), e 4-cumarato:coenzima A ligasi (4CL), che sono necessari per differenziare varie classi di metabolismo secondario fenolico. Tutti questi geni di base codificano importanti enzimi che attivano un certo numero di acidi idrossicinnamici per fornire precursori per la biosintesi di lignine, monolignali, e in effetti tutti gli altri principali metaboliti secondari fenolici nelle piante superiori (Lozoya et al., 1988; Allina et al., 1998; Hu et al., 1998; Ehlting et al., 1999; Lindermayr et al., 2002; Hamberger e Hahlbrock, 2004). Poiché il metabolismo secondario fenolico mostra una considerevole specie-specificità, l’indagine dei geni che codificano gli enzimi biosintetici responsabili sono spesso usati come esempio di chemiotassonomia per comprendere l’evoluzione delle piante. Tuttavia, considerando che l’evoluzione di questi geni in isolamento è piuttosto restrittiva, una comprensione più profonda è fornita combinando questo con l’indagine dell’evoluzione dei geni biosintetici shikimate-fenilalanina nella stirpe verde.
Conclusione
Durante il lungo periodo evolutivo coperto dalle alghe acquatiche alle piante terrestri, le piante si sono adattate alle nicchie ambientali con le strategie evolutive come la duplicazione dei geni e l’evoluzione convergente attraverso il filtraggio della selezione naturale. I geni della biosintesi shikimate delle piante si sono evoluti di conseguenza (Figura 3). In questa revisione, abbiamo dimostrato che i geni biosintetici del metabolismo primario degli aminoacidi aromatici sono ben conservati tra le alghe e tutte le piante terrestri. Tuttavia, in contrasto con le specie di alghe che non hanno né isoforme né geni duplicati nei loro genomi, tutte le piante terrestri ospitano duplicazioni geniche tra cui duplicazioni geniche in tandem che sono particolarmente importanti nei casi di DAHPS, DHQD/SD, CS, CM e ADT (Figura 3A; Tabella 2). La nostra analisi filogenetica ha rivelato una chiara separazione tra alghe, monocotiledoni, dicotiledoni, specie legnose e leguminose. L’analisi della presenza e del numero di copie dei geni chiave in queste specie fornisce diversi suggerimenti su come migliorare la nostra comprensione dell’impalcatura da cui questi geni si sono evoluti. Tuttavia, le esatte pressioni evolutive sui geni della biosintesi shikimate, compresa la presenza unica del complesso Arom, richiederanno ulteriori studi considerevoli. Detto questo è intrigante per confrontare e contrastare i geni biosintetici di quelli a valle di loro nella produzione di fenoli vegetali (Figura 3B). È interessante notare che i geni del percorso shikimate sono onnipresenti in tutta la stirpe verde, mentre questo non può essere detto per tutti i geni a valle della biosintesi dei fenilpropanoidi. Inoltre, c’è una duplicazione genica molto più grande all’interno fenilpropanoide che biosintesi shikimate (Figura 3A; Tabella 2). Questo fatto si riflette anche nel livello di diversità chimica delle rispettive vie, con l’essenzialità della via shikimate che impedisce molta diversità, ma le specie fenilpropanoidi sono spesso ridondanti in funzione l’una dell’altra. Sembrerebbe probabile che il percorso fenilpropanoide inizialmente è sorto tramite mutazioni che si accumulano nei geni del percorso shikimate. Tuttavia, mentre questi erano potenzialmente vantaggiosi nelle piante terrestri per ragioni che discutiamo nella nostra recente revisione di questi composti (Tohge et al., 2013) non sembrano condividere l’essenzialità dello shikimate in tutta la stirpe verde.
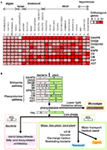
Figura 3. Mappa di calore per le isoforme dei geni biosintetici shikimate-fenilalanina nei genomi delle piante e schema ipotetico per l’evoluzione del metabolismo secondario fenilalanina derivato. (A) Panoramica Heap map del numero di isoforme del gene biosintetico shikimate-fenilalanina in 25 specie. (B) Figura schematica ipotetica per i geni biosintetici shikimate-fenilalanina e la loro evoluzione del metabolismo secondario fenolico.
Conflict of Interest Statement
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Acknowledgments
L’attività di ricerca di Takayuki Tohge è supportata dalla Fondazione Alexander von Humboldt. I finanziamenti della Max-Planck-Society (a Takayuki Tohge, Mutsumi Watanabe, Rainer Hoefgen, Alisdair R. Fernie) sono riconosciuti con gratitudine.
Benesova, M., and Bode, R. (1992). Isoforme di corismato mutasi da semi e piantine di Papaver somniferum. Phytochemistry 31, 2983-2987.
CrossRef Full Text
Fiedler, E., and Schultz, G. (1985). Localizzazione, purificazione e caratterizzazione della shikimate oxidoreductase-deidroquinate hydrolase dallo stroma dei cloroplasti di spinaci. Plant Physiol. 79, 212-218.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Hamberger, B., Ehlting, J., Barbazuk, B., and Douglas, C. J. (2006). Genomica comparativa del percorso shikimate in Arabidopsis, Populus trichocarpa e Oryza sativa: struttura della famiglia di geni del percorso shikimate e identificazione dei candidati per i collegamenti mancanti nella biosintesi della fenilalanina. Recenti Adv. Phytochem. 40, 85-113.
CrossRef Full Text
Maclean, J., and Ali, S. (2003). La struttura della chorismate synthase rivela un nuovo legame di flavina ad una reazione chimica unica. Structure 11, 1499-1511.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Schmid, J., Schaller, A., Leibinger, U., Boll, W., and Amrhein, N. (1992). Il precursore della shikimate kinasi di pomodoro sintetizzato in vitro è enzimaticamente attivo e viene importato e trasformato nell’enzima maturo dai cloroplasti. Plant J. 2, 375-383.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Tzin, V., e Galili, G. (2010). Nuove intuizioni sulle vie di biosintesi di shikimate e aminoacidi aromatici nelle piante. Mol. Plant 3, 956-972.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text