Introducción
La vía del shikimato está estrechamente interrelacionada con las de los aminoácidos aromáticos (L-triptófano, L-fenilalanina y L-tirosina) y en las plantas terrestres soporta flujos muy elevados con estimaciones de la cantidad de carbono fijo que pasa por la vía que varían entre el 20 y el 50% (Weiss, 1986; Corea et al., 2012; Maeda y Dudareva, 2012). La investigación se ha centrado considerablemente en esta vía, ya que los aminoácidos aromáticos no son producidos por los seres humanos y el ganado monogástrico y, por lo tanto, son un componente dietético importante (Tzin y Galili, 2010). Además, una de las enzimas de la vía -la 5-enolpiruvalsimato-3-fosfato sintasa (EPSP)- es una de las dianas de herbicidas más empleadas (véase, Duke y Powles, 2008). Además, como hemos descrito recientemente, los metabolitos secundarios fenólicos de las plantas y sus precursores se sintetizan a través de la vía de la biosíntesis del shikimato y sus numerosos puntos de ramificación (Tohge et al., 2013). La vía del shikimato está muy conservada y se encuentra en hongos, bacterias y especies vegetales, donde opera en la biosíntesis no sólo de los tres aminoácidos aromáticos descritos anteriormente, sino también de innumerables metabolitos secundarios aromáticos como alcaloides, flavonoides, ligninas y antibióticos aromáticos. Muchos de estos compuestos son bioactivos y desempeñan un papel importante en la defensa de las plantas contra el estrés biótico y abiótico y las interacciones con el medio ambiente (Hamberger et al., 2006; Maeda y Dudareva, 2012), por lo que tienen una gran importancia fisiológica. Se estima que, en condiciones normales, hasta el 20% del carbono total fijado fluye a través de la vía del shikimato (Ni et al., 1996), con un mayor flujo de carbono a través de la vía en momentos de estrés de la planta o de crecimiento rápido (Corea et al., 2012). Dada su importancia, quizás no sea sorprendente que todos los miembros de los genes biosintéticos y las correspondientes enzimas implicadas en la vía del shikimato se hayan caracterizado en plantas modelo como Arabidopsis. La comparación entre especies de las enzimas biosintéticas del shikimato ha revelado que comparten similitudes de secuencia, evolución divergente y aspectos comunes en los mecanismos de reacción (Dosselaere y Vanderleyden, 2001). Sin embargo, todas las demás especies varían considerablemente con respecto a los hongos, que han desarrollado un sistema complejo con un único polipéptido pentafuncional conocido como el complejo AroM, que realiza cinco reacciones consecutivas (Lumsden y Coggins, 1977; Duncan et al., 1987). En esta revisión resumiremos los conocimientos actuales sobre la naturaleza genética de esta vía centrándonos en las comparaciones entre especies que abarcan una amplia gama de especies, incluyendo las algas (Chlamydomonas reinhardtii, Volvox carteri, Micromonas sp, Ostreococcus tauri, Ostreococcus lucimarinus), musgos (Selaginella moellendorffii, Physcomitrella patens), monocotiledóneas (Sorghum bicolor, Zea mays, Brachypodium distachyon, Oryza sativa ssp. japonica y Oryza sativa ssp. indica), y dicotiledóneas (Vitis vinifera, Theobroma cacao, Carica papaya, Arabidopsis thaliana, Arabidopsis lyrata, Populus trichocarpa, Ricinus communis, Manihot esculenta, Malus domestica, Fragaria vesca, Glycine max, Lotus japonicus, Medicago truncatula) (Tabla 1). Finalmente, comparamos y contrastamos la evolución de esta vía con la de las vías más especializadas de la biosíntesis de fenilpropanoides.
Tabla 1. Resumen de las especies utilizadas en el estudio.
Biosíntesis de shikimato y metabolismo secundario derivado de la fenilalanina en plantas
Dado que los metabolitos secundarios fenólicos que se derivan de la fenilalanina a través de la biosíntesis de shikimato están ampliamente distribuidos en las plantas y otros eucariotas, los genes que codifican las enzimas de biosíntesis de shikimato están generalmente muy conservados en la naturaleza. En la biosíntesis del shikimato y de la fenilalanina intervienen ocho y dos reacciones, respectivamente. Tanto los miembros de todas las familias de genes como las correspondientes enzimas biosintéticas implicadas en estas vías se han caracterizado en plantas modelo como Arabidopsis (Figura 1A). Por el contrario, los metabolitos secundarios fenólicos derivados de la fenilalanina muestran una considerable distribución específica de las especies, habiéndose encontrado en el reino vegetal metabolitos secundarios fenólicos como los derivados de la cumarina, el monolignal, la lignina, los derivados de la espermidina, el flavonoide y el tanino, presentes en familias específicas dentro del linaje verde (Figura 1B). Esta diversidad ha surgido por la acción de diversas estrategias evolutivas, por ejemplo, la duplicación de genes y la evolución cis-reguladora para adaptarse a las condiciones ambientales imperantes. Dada su distribución específica por especies, los genes implicados en el metabolismo secundario fenólico de las plantas, como la fenilamonioliasa (PAL), la sintasa de policétidos (PKS), las desoxigenasas dependientes de 2-oxoglutarato (2ODDs) y las UDP-glicosiltransferasas (UGTs) se utilizan con frecuencia como casos de estudio de la evolución de las plantas (Tohge et al., 2013). A pesar de que los genes biosintéticos de shikimato-fenilalanina están bien conservados en todas las especies, incluidas las de algas, no se detectaron genes ortólogos relacionados con el metabolismo secundario fenólico en todas las especies de algas (Tabla 2, Tohge et al., 2013). Este resultado sugiere un origen considerablemente más antiguo de las vías del shikimato-fenilalanina. En las siguientes secciones, discutiremos la evolución de las vías del shikimato-fenilalanina centrándonos en comparaciones entre especies para cada gen que codifica una de las enzimas constituyentes de cualquiera de las vías.
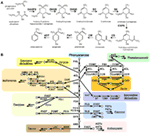
Figura 1. La biosíntesis de metabolitos secundarios derivados del shikimato y la fenilalanina en las plantas. (A) La biosíntesis del shikimato a partir de fosfoenolpiruvato (PEP) y D-eritrosa 4-fosfato se describe con genes caracterizados y metabolitos intermedios reportados. (B) Biosíntesis de mebolitos secundarios fenólicos principales derivados de la fenilalanina en el linaje verde. La flecha indica la reacción enzimática, el círculo indica el metabolito. Abreviatura: DAHPS, 3-deoxi-D-arabino-heptulosonato 7-fosfato sintasa; DQS, 3-dehidroquinato sintasa; DHQD/SD, 3-dehidroquinato deshidratasa; SK, shikimato quinasa; ESPS, 3-fosfoshikimato 1-carboxiviniltransferasa; CS, corismato sintasa; CM, corismato mutasa; PAT, prefenato aminotransferasa; ADT, arogenato deshidratasa. PAL, fenilalanina amonilasa; C4H, cinamato-4-hidroxilasa; 4CL, 4-cumarato CoA ligasa; CAD, cinamoil-alcohol deshidrogenasa; F5H, ferulato 5-hidroxilasa; C3H, cumarato 3-hidroxilasa; ALDH, aldehído deshidrogenasa; CCR, cinamoil-CoA reductasa; HCT, hidroxicinamoil-Coenzima A shikimato/quinato hidroxicinamoiltransferasa; CCoAOMT, cafeoil/CoA-3-O-meteltransferasa; CHS, chalcona sintetizada; CHI, chalcona isomerasa; F3H, flavanona 3-hidroxilasa; F3′H, flavonoide-3′-hidroxilasa; F3GT, flavonoide-3-O-glicosiltransferasa; FS, flavona sintasa; FOMT, flavonoide O-metiltransferasa; FCGT, flavona-C-glicosiltransferasa; FLS, flavonol sintetizado; F3GT, flavonoide-3-O-glicosiltransferasa; DFR, dihidroflavonol reductasa; ANS, antocianidina sintetizada; AGT, flavonoide-O-glicosiltransferasa; AAT, antocianina aciltransferasa; BAN, oxidorreductasa|dihidroflavonol reductasa similar; LAC, lacasa.

Tabla 2. Genes biosintéticos de shikimato y fenilalanina y homólogos en cada especie con/sin genes duplicados en tándem.
3-Deoxi-D-arabino-heptulosonato 7-fosfato sintasa
El primer paso enzimático de la vía del shikimato, la 3-deoxi-D-arabino-heptulosonato 7-fosfato sintasa (DAHPS), cataliza una condensación aldólica de fosfoenolpiruvato (PEP), y D-eritrosa 4-fosfato (E4P) para producir 3-deoxi-D-arabino-heptulosonato 7-fosfato (DAHP) (Figura 1). Según su estructura proteica, las DAHPS pueden agruparse en dos clases de homología distintas. Las DAHPS de clase I derivadas de microbios contienen dominios bifuncionales corismato-mutasa (CM)-DAHPS, por lo que las DAHPS microbianas, por ejemplo, E. coli (AroF, G y H) y S. cerevisiae (Aro3 y 4), se clasifican como DAHPS de clase I. Por el contrario, se pensaba que las DAHPS de clase II sólo estaban presentes en las especies vegetales, pero posteriormente se han notificado en ciertos microbios como Streptomyces coelicolor, Streptomyces rimosus y Neurospora crassa (Bentley, 1990; Maeda y Dudareva, 2012). Sin embargo, las actividades DAHPS (AroA) y CM (AroQ) de la DAHPS de B. subtilis están separadas por el truncamiento del dominio. El análisis detallado de la estructura de la secuencia de las familias bacterianas AroA y AroQ, los estudios enzimáticos con la proteína completa y los dominios truncados de AroA y AroQ de B. subtilis, y la comparación con las proteínas de fusión de Porphyromonas gingivalis en las que el dominio AroQ se fusionó con la terminación C de AroA, sugieren que la «regulación por retroalimentación» puede ser, de hecho, el vínculo evolutivo entre las dos clases que evolucionan a partir de un miembro primitivo no regulado de las DAHPS de clase II (Wu y Woodard, 2006). Las DAHPS de clase II de las plantas se han descrito en las raíces de la zanahoria (Suzich et al., 1985) y en el cultivo de células de patata (Pinto et al., 1986; Herrmann y Weaver, 1999). El DAHPS está codificado por tres genes en el genoma de Arabidopsis (AtDAHPS1, AT4G39980; AtDAHPS2, At4g33510; AtDAHPS3, At1g22410). Las consultas de búsqueda de genes ortólogos utilizando los DAHPS de Arabidopsis, revelaron un único gen en especies de algas (Chlamydomonas reinhardtii, Volvox carteri, Micromonas sp. y Ostreococcus tauri) y Lotus japonica, pero de dos a ocho isoformas en otras especies de plantas superiores (Tabla 2). Los genes de tipo AtDAHPS1 y AtDAHPS2 muestran una expresión diferencial en Arabidopsis thaliana, Solanum lycopersicum y Solanum tuberosum (Maeda y Dudareva, 2012). Los genes de tipo AtDAHPS1, que además están sujetos a la regulación redox por el sistema ferredoxina-tioredoxina, muestran una inducción significativa por las heridas y la infección de patógenos (Keith et al., 1991; Gorlach et al., 1995; Maeda y Dudareva, 2012), mientras que los genes de tipo AtDAHPS2 muestran una expresión constitutiva (Gorlach et al., 1995). Un análisis filogenético de los genes DAHPS revela cuatro clados principales, (i) un clado de microfitas, (ii) un clado de duplicación de briofitas, (iii) un clado de especies leñosas monocotiledóneas y dicotiledóneas, (iv) un clado de AtDAHPSs (Figura 2Aa). Además, el clado mayor iv tiene cuatro subgrupos, (iv-a) grupo AtDAHPS2, (iv-b) monocotiledóneas, (iv-c) grupo AtDAHPS1 y (iv-d) grupo AtDAHP3. Este resultado indica que los genes del tipo AtDAHPS1 de expresión constitutiva y AtDAHPS 3 de respuesta al estrés muestran una secuencia bien conservada entre las especies (clado iv-c y iv-d), mientras que los segundos genes del tipo AtDAHPS2 de expresión constitutiva están claramente separados entre las especies monocotiledóneas y las dicotiledóneas (clado iv-a).

Figura 2. Análisis del árbol filogenético de los genes biosintéticos de shikimato y fenilalanina en 25 especies. Árboles filogenéticos de secuencias de aminoácidos de (A) la vía del shikimato: (a), DAHPS, (b) DHS, (c) DHQD/SD, (d) SK, (e) ESPS, y (f) CS, (B) genes relacionados con la fenilalanina, (a) CM y (b) PAT. Las secuencias de aminoácidos de los genes biosintéticos del shikimato se obtienen de la base de datos Plaza (http://bioinformatics.psb.ugent.be/plaza/). Las relaciones entre las especies consideradas se presentan en el sitio web de Plaza. El árbol filogenético se construyó con las secuencias proteicas alineadas mediante MEGA (versión 5.10; http://www.megasoftware.net/; Kumar et al., 2004) utilizando el método de unión de vecinos con los siguientes parámetros: Corrección de Poisson, deleción completa y bootstrap (1000 réplicas, semilla aleatoria). Las secuencias de proteínas fueron alineadas por Plaza. Los valores en las ramas indican el soporte bootstrap en porcentajes.
3-Dehidroquinato Sintasa
El segundo paso de la vía del shikimato es catalizado por la 3-dehidroquinato sintasa (DHQS), una enzima que promueve el intercambio intramolecular del oxígeno del anillo DAHP con el carbono 7 para convertir el DAHP en 3-dehidroquinato. A diferencia de la situación fúngica detallada anteriormente, el gen DHQS de las plantas es monofuncional y sólo se encuentra como una única copia en todas las especies, con la excepción de Glycine max, que alberga dos genes en su genoma (Figura 2Ab). El análisis filogenético de los genes DHQS revela tres clados principales que consisten en (i) microfitas (ii) briofitas, (iii) monocotiledóneas, (iv) Brassicaceae y (v) especies dicotiledóneas. Intrigantemente, en contraste con otros genes biosintéticos de shikimato, la expresión del gen DHQS no está bien correlacionada con la producción de fenilpropanoides en Arabidopsis (Hamberger et al., 2006).
3-Dehidroquinato deshidratasa/shikimato deshidrogenasa
El 3-Deoxi-D-arabino-heptulosonato 7-fosfato es convertido en 3-dehidroquinato por la enzima bifuncional 3-dehidroquinato deshidratasa/shikimato deshidrogenasa (DHQD/SD), que cataliza, en primer lugar, la deshidratación del DAHP a 3-dehidroquímico y, en consecuencia, la reducción reversible de este intermediario a shikimato utilizando NADPH como cofactor. La DHQD/SD existe en tres formas; shikimato deshidrogenasas específicas de bacterias de clase I (tipo AroE), shikimato/quinato deshidrogenasas de clase II (tipo YdiB), y clase III de shikimato deshidrogenasas (tipo SHD-L) (Michel et al., 2003; Singh et al., 2005). En las plantas de clase IV, la actividad enzimática de la DHQD es 10 veces mayor que la actividad de la SD, lo que indica que la cantidad de 3-dehidrohikimato será más que suficiente para soportar el flujo a través de la vía del shikimato (Fiedler y Schultz, 1985). Esta enzima bifuncional juega un papel importante en la regulación del metabolismo de varias vías metabólicas secundarias fenólicas (Bentley, 1990; Ding et al., 2007). En general, las plantas de semilla contienen un único gen DHQD/SD que contiene una secuencia que codifica un péptido de tránsito plástico en su genoma (Maeda et al., 2011, Tabla 2). Sin embargo, una excepción a esta afirmación es Nicotiana tabacum que contiene dos genes en su genoma. Intrigantemente, el silenciamiento de NtDHD/SHD-1 resulta en una fuerte inhibición del crecimiento y reducción del nivel de aminoácidos aromáticos, ácido clorogénico y contenido de lignina (Ding et al., 2007), sin embargo, una segunda isoforma citosólica puede compensar la producción de shikimato pero no a nivel fenotípico. De forma más general, el análisis filogenético revela que las microfitas también contienen un bajo número de genes DHQD/SD (entre uno y dos), mientras que se pudo observar una clara separación entre (i) el clado de las microfitas, (ii) el clado de las briofitas, (iii) el clado de las monocotiledóneas, (iv) el clado de las especies leñosas con duplicación de genes en tándem, y (v) los clados de las dicotiledóneas (Figura 2Ac; Tabla 2). Curiosamente, la observación del clado de duplicación de genes en tándem específico de las especies leñosas sugiere que estas especies evolucionaron después de la duplicación de genes DHQD/SD. La localización citosólica de NtDHD/SHD-2 es intrigante, ya que se ha informado de la presencia de DAHP sintasa, ESPS sintasa e isoformas de CM que carecen de secuencias N-terminales de orientación plastidial (d’Amato, 1984; Mousdale y Coggins, 1985; Ganson et al., 1986). Además, los hallazgos de que tanto la ESPS sintasa como la shikimato quinasa (SK) están activas incluso cuando conservan sus secuencias objetivo (Dellacioppa et al., 1986; Schmid et al., 1992) sugieren que también podrían ser potencialmente constituyentes de una vía citosólica. Finalmente, los experimentos en los que se suministraron mitocondrias aisladas y altamente puras con glucosa marcada con 13C para investigar la unión de las isoformas citosólicas de la glucólisis (Giege et al., 2003) también revelaron un enriquecimiento de 13C en el shikimato (Sweetlove y Fernie, 2013), lo que indica que es probable que exista una vía citosólica completa también en esta especie.
Shikimato quinasa
La quinta reacción de la vía del shikimato es catalizada por la SK, que cataliza la fosforilación dependiente de ATP del shikimato a shikimato 3-fospato (S3P). E. coli tiene dos SKs, una de clase I (tipo AroL) y otra de II (tipo AroK) que comparten sólo un 30% de identidad de secuencia (Griffin y Gasson, 1995; Whipp y Pittard, 1995; Herrmann y Weaver, 1999). En las plantas, se encuentran diferentes números de isoformas SK en varias especies; sólo una en algas verdes, licófitas y briofitas, pero entre una y tres en plantas monocotiledóneas y dicotiledóneas (Tabla 2). Un análisis filogenético de los genes SK presenta cinco clados principales que consisten en (i) microfito, (ii) briófito, (iii) clado específico de especies leñosas dicotiledóneas, (iv) clado de monocotiledóneas y (v) clado de especies dicotiledóneas (Figura 2Ad). El análisis de la proteína SK de Spinacia olerancea reveló que estaba modulada por el estado energético y, por tanto, es similar a la proteína SK bacteriana y a otras enzimas que utilizan ATP (Pacold y Anderson, 1973; Huang et al., 1975; Schmidt et al., 1990). Por esta razón, recientemente se ha postulado que SK puede vincularse a la vía del shikimato que requiere energía para el equilibrio energético celular (Maeda y Dudareva, 2012), sin embargo, actualmente se carece de apoyo experimental directo para esta hipótesis. En Arabidopsis, se ha demostrado que los genes homólogos llamados SKL1 y SKL2, que son funcionalmente necesarios para la biogénesis del cloroplasto, han surgido de la duplicación del gen SK (Fucile et al., 2008). Se han encontrado ortólogos de SKL1 y SKL2 en varias especies de plantas de semilla, pero no en algas verdes (Tabla 2).
5-enolipiruvilshikimato 3-fosfato sintasa
La 5-enolipiruvilshikimato 3-fosfato sintasa (EPSPS, 3-fosfoshikimato 1-carboxitransferasa) es el sexto paso y aquí se condensa una segunda PEP con S3P para formar 5-enolpiruvilshikimato 3-fosfato (EPSP). Dado que la EPSPS es el único objetivo conocido para el herbicida glifosato (Steinrucken y Amrhein, 1980), las isoformas de esta enzima se clasifican a menudo de acuerdo con su sensibilidad al glifosato, la EPSPS sensible al glifosato de clase I está presente en bacterias y especies de plantas, mientras que la EPSPS insensible al glifosato de clase II que se ha reportado en ciertas bacterias como Agrobacterium (Fucile et al., 2011). En las plantas, se encuentra un número diferente de isoformas EPSPS en varias especies; una sola isoforma en las algas verdes, licófitas y briofitas, pero una o dos se encuentran en las especies monocotiledóneas y dicotiledóneas (Tabla 2). El análisis filogenético de los genes EPSPS reveló, de forma atípica para los genes asociados al metabolismo del shikimato, que se podían observar cinco grupos principales: (i) microfitos, (ii) briofitos, (iii) clado específico de Brassicaceae, (iv) especies monocotiledóneas y (v) clado de especies dicotiledóneas (Figura 2Ae). Existen claros indicios de que los genes EPSPS duplicados en Arabidopsis, manzana, vid, soja y álamo son el resultado de eventos de duplicación independientes dentro de sus linajes, manteniéndose ambas copias en Arabidopsis (Hamberger et al., 2006), sin embargo, la razón de la divergencia única en este gen de la vía no está actualmente clara.
Corismato sintasa
El corismato, el producto final de la vía del shikimato, se forma posteriormente por la corismato sintasa (CS) que cataliza la eliminación trans-1,4 del fosfato de la EPSP. Las CSs se clasifican dentro de uno de los dos grupos funcionales (i) CSs bifuncionales de tipo fúngico que se asocian con la flavin reductasa dependiente de NADPH o (ii) CSs monofuncionales de tipo bacteriano y vegetal (Schaller et al., 1991; Maeda y Dudareva, 2012). La reacción catalizada por la CS requiere mononucleótido de flavina (FMN) y su reacción global es redox neutral (Ramjee et al., 1991; Macheroux et al., 1999; Maclean y Ali, 2003). El FMN representa un donante de electrones para la EPSP que facilita la escisión del fosfato. El primer gen CS clonado de una planta fue el de C. sempervirens (Schaller et al., 1991) que contiene un único CS en su genoma. Dado que este gen tiene una secuencia de señal de importación de plástidos en los 5′, estos resultados indican que puede no haber CS fuera del plástido en esta especie. El estudio de otras especies reveló que entre uno y dos genes CS estaban presentes en algas verdes, licófitas y briofitas, así como en especies dicotiledóneas, pero que entre uno y tres están presentes en los genomas de especies de manzana y leguminosas (Tabla 2). Un análisis filogenético de los genes CS revela tres clados principales constituidos por (i) microfitas, (ii) monocotiledóneas, (iii) especies dicotiledóneas (Figura 2Af).
Corismato Mutasa
La corismato mutasa cataliza el primer paso de la biosíntesis de la fenilalanina y la tirosina y además representa un paso clave hacia la división de la rama de la biosíntesis del triptófano. La CM cataliza la transformación de corismato en prefenato mediante un reordenamiento de Claisen. Las proteínas CM menores bacterianas (tipo AroQ, clase I CM) muestran una actividad enzimática monofuncional mientras que varias CMs bifuncionales como CM-PDT, CM-PDH, y CM-DAHP se han encontrado adicionalmente en hongos y bacterias (clase II CM, Euverink et al., 1995; Romero et al., 1995; Chen et al., 2003; Baez-Viveros et al., 2004). A pesar del hecho de que sólo un gen CM está presente en los genomas de algas y licófitos, más una copia del gen (dos a cinco) se encuentran en las briofitas, así como en las especies monocotiledóneas y dicotiledóneas (Tabla 2). En las plantas de semilla, el CM1 lleva un péptido putativo de tránsito plastidial, pero el CM2 no y además suele ser insensible a la regulación alostérica por aminoácidos aromáticos (Benesova y Bode, 1992; Eberhard et al., 1996; Maeda y Dudareva, 2012). Varias especies de plantas, especialmente las dicotiledóneas, tienen un gen adicional de la familia CM3 que muestra una alta similitud de secuencia con el CM2, pero que lleva un péptido putativo de tránsito plastidial. Por ejemplo, Arabidopsis tiene tres isozimas llamadas AtCM1 (At3g29200), AtCM2 (At5g10870) y AtCM3 (At1g69370) (Mobley et al., 1999; Tzin y Galili, 2010). El análisis filogenético de los genes CS revela tres clados principales constituidos por (i) el clado AtCM2, (ii) el clado de microfitas y briofitas, y (iii) el clado AtCM2 (Figura 2Ba). Además, el clado iii muestra dos subgrupos, (iii-a) el subgrupo AtCM3 y (iii-b) el subgrupo AtCM1 (Figura 2Ba) (Eberhard et al., 1996). A pesar de que el subgrupo CM2 contiene todas las especies de plantas de semilla, las especies monocotiledóneas no están contenidas en el subgrupo AtCM3. Recientemente la importancia del CM se ha extendido más allá del metabolismo intracelular, En Zea mays, la corismato mutasa Cmu1 secretada por Ustilago maydis, un patógeno muy extendido caracterizado por el desarrollo de grandes tumores en las plantas y comúnmente conocido como tizón, es un factor de virulencia. La captación de la proteína CMu1 de Ustilago por parte de las células vegetales permite redirigir el metabolismo de la planta y cambiar el estado metabólico de estas células mediante el cebado metabólico (Djamei et al., 2011). Ahora parece que las CMs secretadas se encuentran en muchos microbios relacionados con las plantas y esta forma de manipulación del huésped parece ser un arma general en el arsenal de los patógenos de las plantas.
Prefenato Aminotransferasa y Arogenato Deshidratasa
La fenato aminotransferasa (PAT) y la arogenato deshidratasa (ADT) catalizan los pasos finales para la producción de fenilalanina. Mientras que la ADT se clonó por primera vez en 2007 (Cho et al., 2007; Huang et al., 2010), sólo recientemente se ha clonado la PAT. Los trabajos publicados en 2011 identificaron PAT en el híbrido de Petunia, Arabidopsis thaliana y Solanum lycopersicum (Dal Cin et al., 2011; Maeda et al., 2011) y establecieron que dirige el flujo de carbono del prefenato al arogenato, pero también que está fuerte y coordinadamente regulado con los genes del metabolismo primario y los volátiles del sabor derivados de la fenilalanina. En las especies vegetales se ha encontrado un número diferente de isoformas PAT. Aunque las algas verdes sólo contienen genes PAT y ADT únicos, las especies monocotiledóneas tienen entre una y dos PAT y entre dos y cuatro ADT, mientras que los genomas de las plantas dicotiledóneas contienen el mismo número de PAT pero de dos a ocho ADT (Tabla 2). El análisis filogenético de los genes PAT muestra tres clados principales de (i) especies microfitas, (ii) monocotiledóneas y (iii) dicotiledóneas (Figura 2Bb).
Genes implicados en los metabolismos secundarios fenólicos de las plantas
El metabolismo secundario fenólico muestra una inmensa diversidad química debido a la evolución de los genes enzimáticos que intervienen en las distintas vías biosintéticas y decorativas. Dicha variación se debe a la diversidad y redundancia de varios genes clave del metabolismo secundario fenólico, como las PKS, los citocromos P450 (CYP), las dioxigenasas dependientes de Fe2+/2-oxoglutarato (2ODD) y las UDP-glicosiltransferasas (UGT). Por otro lado, hay otros genes biosintéticos generales relacionados con los fenilpropanoides, la fenilalanina amoníaco-liasa (PAL), la cinamato 4-hidroxilasa (C4H) y la 4-cumarato:coenzima A ligasa (4CL), que son necesarios para diferenciar varias clases de metabolismo secundario fenólico. Todos estos genes centrales codifican importantes enzimas que activan una serie de ácidos hidroxicinámicos para proporcionar precursores para la biosíntesis de ligninas, monolignales y, de hecho, todos los demás metabolitos secundarios fenólicos principales en plantas superiores (Lozoya et al., 1988; Allina et al., 1998; Hu et al., 1998; Ehlting et al., 1999; Lindermayr et al., 2002; Hamberger y Hahlbrock, 2004). Dado que el metabolismo secundario fenólico muestra una considerable especificidad de especie, la investigación de los genes que codifican las enzimas biosintéticas responsables se utiliza con frecuencia como ejemplo de quimiotaxonomía para entender la evolución de las plantas. Sin embargo, considerando que la evolución de estos genes de forma aislada es bastante restrictiva, se proporciona una comprensión más profunda combinando esto con la investigación de la evolución de los genes biosintéticos de shikimato-fenilalanina en el linaje verde.
Conclusión
Durante el largo período evolutivo que abarca desde las algas acuáticas hasta las plantas terrestres, las plantas se han adaptado a los nichos ambientales con las estrategias evolutivas como la duplicación de genes y la evolución convergente por la filtración de la selección natural. Los genes de la biosíntesis del shikimato en las plantas han evolucionado en consecuencia (Figura 3). En esta revisión, hemos demostrado que los genes biosintéticos del metabolismo primario de los aminoácidos aromáticos están bien conservados entre las algas y todas las plantas terrestres. Sin embargo, en contraste con las especies de algas que no tienen ni isoformas ni genes duplicados en sus genomas, todas las plantas terrestres albergan duplicaciones de genes, incluyendo duplicaciones de genes en tándem que son particularmente prominentes en los casos de DAHPS, DHQD/SD, CS, CM y ADT (Figura 3A; Tabla 2). Nuestro análisis filogenético reveló una clara separación entre algas, monocotiledóneas, dicotiledóneas, especies leñosas y leguminosas. El análisis de la presencia y el número de copias de los genes clave en estas especies ofrece varias pistas para mejorar nuestra comprensión del andamiaje a partir del cual han evolucionado estos genes. Sin embargo, las presiones evolutivas exactas sobre los genes de la biosíntesis del shikimato, incluida la presencia única del complejo Arom, requerirán considerables estudios adicionales. Dicho esto, es intrigante comparar y contrastar los genes de la biosíntesis de aquellos que se encuentran aguas abajo de ellos en la producción de fenólicos de las plantas (Figura 3B). Curiosamente, los genes de la vía del shikimato son ubicuos en todo el linaje verde, mientras que esto no puede decirse de todos los genes posteriores de la biosíntesis de fenilpropanoides. Además, hay una duplicación de genes mucho mayor dentro de la biosíntesis de fenilpropanoides que de shikimatos (Figura 3A; Tabla 2). Este hecho también se refleja en el nivel de diversidad química de las respectivas vías, con la esencialidad de la vía del shikimato impidiendo mucha diversidad, pero las especies de fenilpropanoides a menudo son redundantes en función unas de otras. Parece probable que la vía de los fenilpropanoides surgiera inicialmente a través de mutaciones acumuladas en los genes de la vía del shikimato. Sin embargo, aunque estos fueron potencialmente beneficiosos en las plantas terrestres por las razones que discutimos en nuestra reciente revisión de estos compuestos (Tohge et al., 2013) no parecen compartir la esencialidad del shikimato en todo el linaje verde.
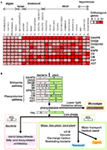
Figura 3. Mapa de calor para las isoformas de los genes biosintéticos de shikimato-fenilalanina en los genomas de las plantas y esquema hipotético para la evolución del metabolismo secundario fenólico derivado de la fenilalanina. (A) Mapa del número de isoformas de genes biosintéticos de shikimato-fenilalanina en 25 especies. (B) Figura esquemática hipotética de los genes biosintéticos de shikimato-fenilalanina y su evolución del metabolismo secundario fenólico.
Declaración de conflicto de intereses
Los autores declaran que la investigación se ha llevado a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial conflicto de intereses.
Agradecimientos
La actividad investigadora de Takayuki Tohge está apoyada por la Fundación Alexander von Humboldt. Se agradece la financiación de la Sociedad Max-Planck (a Takayuki Tohge, Mutsumi Watanabe, Rainer Hoefgen, Alisdair R. Fernie).
Benesova, M., y Bode, R. (1992). Chorismate mutase isoforms from seeds and seedlings of Papaver somniferum. Phytochemistry 31, 2983-2987.
CrossRef Full Text
Fiedler, E., and Schultz, G. (1985). Localization, purification, and characterization of shikimate oxidoreductase-dehydroquinate hydrolase from stroma of spinach-chloroplasts. Plant Physiol. 79, 212-218.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Hamberger, B., Ehlting, J., Barbazuk, B., and Douglas, C. J. (2006). Genómica comparativa de la vía del shikimato en Arabidopsis, Populus trichocarpa y Oryza sativa: estructura de la familia de genes de la vía del shikimato e identificación de candidatos a eslabones perdidos en la biosíntesis de la fenilalanina. Recent Adv. Phytochem. 40, 85-113.
CrossRef Full Text
Maclean, J., and Ali, S. (2003). La estructura de la corismato sintasa revela una nueva unión de flavina a una reacción química única. Structure 11, 1499-1511.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Schmid, J., Schaller, A., Leibinger, U., Boll, W., y Amrhein, N. (1992). El precursor de la shikimato quinasa de tomate sintetizado in vitro es enzimáticamente activo y es importado y procesado a la enzima madura por los cloroplastos. Plant J. 2, 375-383.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text
Tzin, V., and Galili, G. (2010). New insights into the shikimate and aromatic amino acids biosynthesis pathways in plants. Mol. Plant 3, 956-972.
Pubmed Abstract | Pubmed Full Text | CrossRef Full Text