Int J Biol Sci 2008; 4(6):338-344. doi:10.7150/ijbs.4.338
Review
Celina Janion ![]()
Istituto di Biochimica e Biofisica, Accademia Polacca delle Scienze, Pawinskiego 5a, 02-106 Warszawa, Polonia
Janion C. Inducible SOS risposta sistema di riparazione del DNA e mutagenesi in Escherichia coli. Int J Biol Sci 2008; 4(6):338-344. doi:10.7150/ijbs.4.338. Disponibile da https://www.ijbs.com/v04p0338.htm
Il DNA cromosomico è esposto a continui danni e riparazioni. Le cellule contengono una serie di proteine e sistemi di riparazione del DNA specifici che aiutano a mantenere la sua struttura corretta. La risposta SOS è stato il primo sistema di riparazione del DNA descritto in Escherichia coli, indotto al trattamento dei batteri con agenti dannosi per il DNA che arrestano la replicazione del DNA e la divisione cellulare. L’induzione della risposta SOS coinvolge più di quaranta geni SOS indipendenti, la maggior parte dei quali codifica proteine impegnate nella protezione, riparazione, replicazione, mutagenesi e metabolismo del DNA. In condizioni di crescita normali i geni SOS sono espressi a un livello basale, che aumenta distintamente all’induzione della risposta SOS. La risposta SOS è stata trovata in molte specie batteriche (ad esempio, Salmonella typhimurium, Caulobacter crescentus, Mycobacterium tuberculosis), ma non in cellule eucariotiche. Tuttavia, le specie di tutti i regni contengono alcune proteine SOS-simili che partecipano alla riparazione del DNA e che presentano un’omologia aminoacidica e attività enzimatiche simili a quelle trovate in E. coli, ma non sono organizzate in un sistema SOS. Questo articolo presenta una breve rassegna aggiornata che descrive la scoperta del sistema SOS, la fisiologia dell’induzione SOS, i metodi per la sua determinazione e il ruolo di alcuni geni indotti da SOS.
Keywords: SOS response, DNA repair, DNA mutations, error-prone repair, mutagenic DNA polymerases
Historical Overview
Dopo che è stato riconosciuto che i geni sono composti da DNA (Oswald T. Avery, 1940), sono stati fatti numerosi esperimenti per esplorare le proprietà chimiche del DNA, soprattutto trattando batteri e batteriofagi con una varietà di agenti e sostanze chimiche, come la luce UV, la mitomicina C (MC), ecc. Di conseguenza, è stato ottenuto un elenco crescente di mutanti batterici che mostrano proprietà nuove e insolite, e le loro proprietà sono state successivamente determinate.
L’ipotesi della risposta SOS è stata sviluppata sulla base dei seguenti dati: (i) L’osservazione di Jean Weigle nel l953 che la riattivazione del fago λ irradiato dai raggi UV aumentava notevolmente quando i fagi irradiati venivano piastrati su cellule ospiti di E. coli precedentemente irradiate. Questo fenomeno è stato in seguito chiamato W- o Weigle- riattivazione; (ii) Induzione del fago λ e lisi dei batteri (trasformazione da sviluppo lisogeno a litico) quando i lisogeni batterici di E. coli sono stati irradiati UV, e (iii) Osservazione della crescita filamentosa delle cellule B di E. coli in risposta all’irradiazione UV, suggerendo una relazione tra l’arresto della divisione cellulare, i meccanismi di induzione del fago λ e la mutazione indotta dagli UV. . Questi dati portarono Miroslav Radman a concludere che in E. coli esiste un sistema di riparazione del DNA dipendente dalle proteine LexA e RecA che viene indotto quando il DNA è gravemente danneggiato e la sua sintesi arrestata e l’induzione di questo sistema è collegata all’induzione di mutazioni. Radman lo chiamò “SOS repair” e “SOS replication” dal nome di un segnale di soccorso telegrafico (o ottico) internazionale “SOS” in alfabeto Morse (tre punti, tre trattini, tre punti).
L’ipotesi SOS di Miroslav Radman fu inizialmente avanzata in una lettera inedita inviata a numerosi ricercatori nel l970, che fu poi pubblicata solo nel 1974 . Evelyn Witkin aveva ipotizzato in precedenza che la formazione di filamenti e l’induzione di profagi nelle cellule B di E. coli irradiate potessero avere un meccanismo correlato. La lettera originale di Radman e il primo articolo di Witkin, considerati la base per la scoperta del fenomeno della risposta SOS, sono stati recentemente ristampati in un articolo di Bryn A. Bridges. Ulteriori lavori su questa linea hanno confermato e sviluppato questa ipotesi. Sistemi che assomigliano in qualche modo alla risposta SOS descritta in E. coli sono stati successivamente trovati per operare anche in cellule eucariotiche, ma le risposte batteriche ed eucariotiche sono in realtà sostanzialmente diverse.
Meccanismo di induzione SOS: Ruolo della coproteasi RecA* e della proteina repressore LexA
I geni recA e lexA sono stati i primi ad essere riconosciuti come coinvolti nell’induzione di SOS. Le mutazioni in questi geni rendono le cellule altamente sensibili alle radiazioni UV. Le proteine LexA da 27 kDa e RecA da 36 kDa erano precedentemente note come proteine di ricombinazione che operano nella vita sessuale e nello scambio genetico dei batteri. Attualmente, è noto che la proteina RecA partecipa anche allo scambio genetico del DNA, alla riparazione del DNA ricombinante recF, recO, recR, recN e ruvABC-dipendente e, insieme alla proteina LexA, svolge un ruolo importante nella regolazione della risposta SOS. La down- e up-regulation dei geni indotti da SOS è fondamentalmente un’interazione di due proteine, LexA repressore e RecA* dove LexA è una proteina repressore trascrizionale, e RecA* è una coproteasi che aiuta l’autocleavage autocatalitico di LexA.
Agenti capaci di indurre il sistema di risposta SOS sono, per esempio, raggi UV, MC, metil metano sulfonato (MMS), e molte altre sostanze chimiche che distruggono il DNA, arrestano la sintesi del DNA e la divisione cellulare, e portano all’accumulo di DNA a singolo filamento (ss). Il livello della proteina RecA nelle cellule batteriche (come quello dell’elicasi II di UvrD) è molto alto. La proteina RecA ha una forte tendenza a formare filamenti nucleoproteici su ssDNA, e una molto più debole con DNA rotto, a doppio filamento (ds). Questo probabilmente protegge il DNA dalla distruzione ed è richiesto per ogni aspetto dell’attività di RecA. L’assemblaggio di RecA su ssDNA procede in direzione 5′-3′ in un rapporto di 1 molecola RecA per 3 basi di DNA, e richiede dATP o ATP, ma nessuna attività ATP-asi. Il disassemblaggio, al contrario, richiede l’idrolisi di ATP in ADP e procede molto più lentamente dell’assemblaggio. RecA assemblato su ssDNA acquisisce un’attività coproteasica, RecA*, che facilita l’auto-cleavage della proteina LexA con conseguente derepressione dei geni regolati da SOS. La proteina LexA ha una debole attività di auto-cleavage, ma la sua scissione e la derepressione dei geni SOS avvengono solo in presenza della coproteasi RecA*.
Ognuno dei geni SOS-inducibili al danno (din) o sos ha vicino al suo sito promotore/operatore una specifica “SOS-box” lunga 20-nucleotidi (chiamata anche LexA-box) alla quale si lega la proteina repressore LexA, impedendo il legame della RNA polimerasi e l’espressione del gene. La scatola SOS ha una struttura palindromica che suggerisce che il repressore LexA si lega come un dimero, come è stato successivamente confermato. Il ruolo della coproteasi RecA* nelle cellule indotte da SOS è quindi: 1. assistere nella scissione della proteina LexA (202 aminoacidi) nel sito Ala84-Gly85, che causa la derepressione dei geni SOS; 2. 2. scindere il repressore CI del fago λ lambda, che trasforma il fago da una forma lisogena a una litica; 3. elaborare UmuD → UmuD’ intaccando UmuD al sito Cys24-Gly25 che è un prerequisito per l’assemblaggio della DNA polimerasi V (Pol V) mutagena indotta da SOS, costituita da UmuD’2C. Il passo limitante della sintesi di Pol V è il processamento UmuD→ UmuD’, che avviene molto più lentamente dell’auto-cleavage di LexA. Il ruolo di Pol V nella mutagenesi è la sintesi del translesione (TLS) attraverso il danno nel DNA modello, permettendo la replicazione del DNA, spesso a costo della fedeltà che porta alla mutazione. Tutte queste proteine, il repressore CI del fago λ e il repressore LexA, UmuD, le proteine PolB/DinA (Pol II) e DinB (Pol IV) sono omologhe nei loro domini carbossi-terminali, e tutte sono codificate dai geni din (sos) regolati in risposta SOS.
L’induzione della risposta SOS procede fino a 45-60 min dopo il trattamento dei batteri con agenti induttori SOS e poi cessa bruscamente. Entro questo tempo la maggior parte delle lesioni sono state riparate. La tempistica della derepressione dei singoli geni din dipende dalla forza del legame del repressore LexA con la scatola SOS e dalla facilità con cui il repressore LexA si stacca da una particolare scatola SOS.
3.1. La risposta SOS è stata studiata in precedenza testando l’aumento dell’espressione dei geni din o dai geni naturali, o utilizzando un gene reporter, ad esempio la fusione di un promotore din putativo con il gene lacZ senza promotore che codifica la β-galattosidasi. Graham Walker e colleghi sono stati i primi a impiegare per questo compito un fago difettoso, Mu1d(Ap,lacZ) costruito da Casadaban e Cohen, che si inserisce facilmente in modo casuale nel cromosoma di E. coli K12 e crea una mutazione. Questo fago porta un gene lacZ senza promotore, per cui la β-galattosidasi non viene espressa. Tuttavia, quando il fago Mu viene casualmente integrato sotto il promotore di un gene din formando un operone funzionale din::Mu-1d(Ap,lacZ), la β-galattosidasi viene sintetizzata in risposta al danno al DNA.
Siccome l’idrolisi del substrato della β-galattosidasi (o-nitrofenil-β-galattoside) forma colonie gialle, quelle che portano una fusione din::Mu-lacZ sono facilmente selezionate su piastre di agar; il livello preciso di β-galattosidasi espresso in risposta al danno al DNA può quindi essere accuratamente misurato in mezzo liquido (vedi Fig.1 per i dettagli). In questo modo sono stati individuati più di dieci nuovi din-genes e la maggior parte di essi sono stati successivamente identificati.
Cinetiche di induzione della β-galattosidasi in ceppi di fusione din::lacZ da parte della mitomicina C (MC). Le fusioni din::lac sono state generate dall’inserimento del batteriofago Mu d1(Ap, lac) nel cromosoma di E. coli. Il derivato λ::Mu d1(Ap lac) è stato generato da un inserimento di Mu d1(Ap lac) del fago λ nel cromosoma di E. coli. Simboli: o, ceppo di fusione non trattato; ●, ceppi di fusione più MC; lexA(Ind-) derivati del ceppo di fusione più MC; ■, recA (Def) derivati del ceppo di fusione più MC; ▼, un derivato contenente pKM280 del del λ:: Mu d1(Ap lac) ceppo più MC. Ristampato da con il permesso dell’autore. Due dei geni, dinA e dinB sono stati successivamente identificati come polB (Pol II) e Pol IV, rispettivamente.
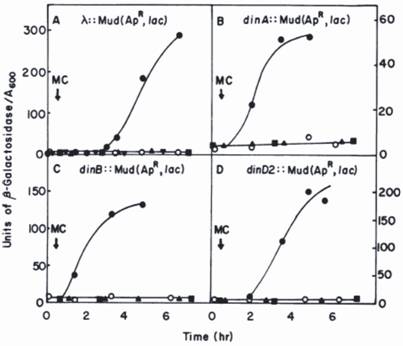 (Clicca sull’immagine per ingrandirla.)
(Clicca sull’immagine per ingrandirla.)
Di recente, un nuovo metodo è stato elaborato per misurare l’espressione del gene SOS e l’attività del promotore dei geni SOS (ad es, recA, lexA, umuDC) utilizzando un plasmide che porta un promotore SOS da studiare fuso al gene reporter gfp che codifica la proteina fluorescente verde (GFP). Questo permette di misurare l’attività del promotore dei geni SOS in una singola cellula batterica, così come la localizzazione e la durata dell’induzione SOS. Sembra che l’induzione dei geni SOS non proceda come un singolo evento, ma segua in diversi passi ripetibili la cui modulazione dipende dalla dose di induzione SOS, dal livello di danno nel DNA e dall’accumulo della proteina UmuD’. Questo metodo apre un nuovo modo di misurare la dinamica della risposta SOS.
3.2. Con la ricerca delle caselle SOS: Determinazione dell’indice di eterologia (HI)
I progressi nel sequenziamento del DNA e la conoscenza degli elementi caratteristici delle sequenze di geni SOS hanno permesso la ricerca computazionale diretta dei geni SOS-riducibili. Quando il 33% del DNA cromosomico di E. coli è stato sequenziato, Lewis et al. hanno localizzato tramite analisi di sequenza ed esperimenti quantitativi di legame al DNA sei nuovi geni potenzialmente regolati da LexA, e li hanno chiamati sosA-F . Per due di questi, sosC e sosD, gli autori hanno confermato sperimentalmente che hanno fortemente legato il repressore LexA purificato.
Successivamente, confrontando le sequenze delle SOS-box di 19 geni din noti all’epoca (compresi sosA-sosF), stabilirono che la sequenza di consenso delle SOS box è un palindromo perfetto, TACTG(TA)5CAGTA ; e sulla base della teoria di Berg e von Hippel calcolarono matematicamente per ciascuna delle SOS-box un indice di eterologia (HI). Questo indice indica la deviazione di una casella SOS dal consenso e, quando il suo valore è basso il gene è strettamente soppresso, e quando il suo valore è alto è più facilmente de-represso. Con un HI maggiore di 15 il repressore LexA non si lega alla SOS-box. Quindi, il valore HI è una misura della forza relativa del legame del repressore LexA a una data casella SOS, ed è responsabile della variazione del potenziale di derepressione.
Quando la sequenza del DNA dell’intero cromosoma di E. coli K12 è stata determinata, Fernandez de Henestrosa et al hanno localizzato, cercando potenziali SOS-box associate a strutture di lettura aperte, 69 potenziali SOS-box con un valore HI ≤ 15, compresi tutti quelli precedentemente noti e sette nuovi. I nuovi geni sono stati successivamente analizzati per la loro capacità di essere espressi su trattamento MC, per la lunghezza del mRNA espresso, e i valori HI (Fig. 2). Le analisi sono state condotte in tre ceppi isogenici di E. coli che differiscono per l’allele lexA: lexA+ (wild type) SOS-inducibile, SOS non-inducibile lexA3(Ind-), e l’allele lexA51(Def) costitutivamente espresso. Le potenziali funzioni dei nuovi e vecchi geni sono state ulteriormente caratterizzate e discusse. I risultati hanno confermato che ognuno dei nuovi geni contenenti SOS box era effettivamente un gene LexA-dipendente, e la sua espressione è stata indotta da MC solo nel ceppo lexA+; altrimenti, erano o non espressi (lexA3), o completamente espressi (lexA51), indipendentemente dal fatto che i batteri fossero trattati con MC o meno (vedi Fig 2 per i dettagli).
Dalla sequenza nucleotidica del gene ydjQ (nomi alternativi b1741 sosD) è stato dedotto che esso codifica la proteina lunga 295 aminoacidi che condivide una significativa omologia con la metà N -terminale della proteina UvrC. La UvrABC-escinucleasi (composta dalle proteine UvrA, UvrB e UvrC) era nota per essere coinvolta nella riparazione dell’escissione nucleotidica (NER) che rimuove addotti voluminosi o lesioni che alterano la struttura (ad esempio, dimeri di pirimidina, pirimidina (6-4) fotoprodotti, e siti abasici) dal DNA modificato. Era anche noto che la parte N di UvrC incide il ssDNA inizialmente sul lato 3′ della lesione, e poi la parte C di UvrC incide il DNA sul lato 5′ della lesione. Recentemente, Moolenaar et al, hanno rinominato la proteina YdjQ come Cho (dopo l’omologo di UvrC) e hanno esaminato la sua attività enzimatica. Hanno confermato che la proteina Cho incide i complessi di preincisione UvrB-DNA solo sul lato 3′ della lesione; tuttavia, alcune lesioni nel DNA che sono state incise molto male dalla proteina UvrC sono state incise molto efficacemente dalla proteina Cho. Quindi, la proteina Cho aumenta notevolmente la gamma di substrati nella riparazione del DNA da parte del sistema NER. I geni uvrA, uvrB e ydjQ (ma non uvrC) sono geni indotti da SOS.
Analisi nordica dei geni di E. coli che sembrano essere regolati da LexA. L’RNA è stato estratto da tre ceppi isogenici che differivano per il gene LexA: RW118 (lexA+), RW434 (lexA) e RW542 (lexA51). L’RNA è stato ottenuto da cellule non danneggiate (-) e da cellule che erano state esposte alla mitomicina C (5 µg ml-1) (+) per 30 minuti prima dell’estrazione. I geni recA e umuDC precedentemente identificati e regolati da LexA sono stati usati come controlli positivi. I geni sono rappresentati secondo il loro indice di eterologia ascendente (HI). Le dimensioni del trascritto mRNA e le possibili funzioni dei geni sono anche indicati Vedi rif. per maggiori dettagli. (Per gentile concessione di Blackwell Science).
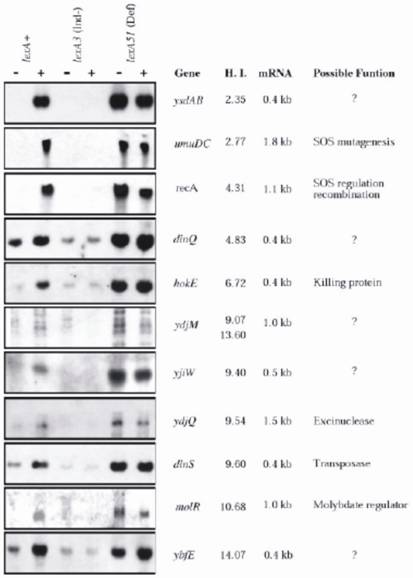 (Clicca sull’immagine per ingrandirla.)
(Clicca sull’immagine per ingrandirla.)
3.3. Localizzazione dei geni din mediante tecniche di microarray
La tecnica del microarray permette di monitorare un gran numero di geni in un esperimento. Courcelle et al., hanno usato microarray contenenti frammenti cromosomici di DNA di E coli amplificati con cornici di lettura aperte di 4101 geni (95,5% del totale) per misurare l’espressione di tutti i geni in ceppi SOS-inducibili (lexA+) e non-inducibili lexA1(Ind-) irradiati UV e non irradiati. Nel ceppo lexA+ irradiato dagli UV gli autori hanno identificato 17 nuovi geni indotti da SOS dipendenti da LexA, oltre ai 26 noti in precedenza; quindi il numero totale di geni indotti da SOS in E. coli è probabilmente 43. Nella stessa pubblicazione gli autori hanno stabilito che il gene ssb che codifica per una proteina legante ssDNA non è SOS-inducibile, come si pensava in precedenza. Hanno anche osservato un certo numero di geni la cui espressione è aumentata (di solito non più di due volte) nelle cellule irradiate dai raggi UV, ma che non erano regolati dalla proteina LexA. Hanno notato che i trascritti proteici di molti geni non regolati da LexA erano ridotti dopo l’irradiazione UV, e hanno concluso che questi trascritti erano probabilmente danneggiati o degradati dagli UV. Hanno anche identificato trenta geni con potenziali strutture simili a SOS box, ma che non erano regolati da LexA.
Meccanismo e specificità del legame del repressore LexA alle SOS box
Si ritiene che siano state identificate le sequenze di tutte le potenziali SOS box nel cromosoma di E. coli. Alcuni esempi di scatole SOS che legano (A) o non legano (B e C) il repressore LexA, insieme ai valori HI e al numero di mismatch (NM) sono mostrati nella tabella 1. NM denota il numero di posizioni nelle caselle SOS che si discostano da un palindromo perfetto. Sia il numero che il modello dei mismatches possono essere la chiave per la specificità del legame della proteina LexA ad ogni singola casella SOS. Sembra che questa ipotesi sia una buona spiegazione per la specificità e la diversa forza del legame della proteina LexA alle sequenze di scatole SOS. Ma questo dovrebbe essere confermato.
Si può vedere che generalmente, quando le SOS-box hanno bassi valori di HI, tra 2,7 e 12, possono legare il repressore LexA (Tabella 1A); e quando l’HI è superiore a 16,4 (Tabella 1B) apparentemente non sono in grado di legare il repressore LexA. Tuttavia, in alcuni casi (mostrati nella parte C) come i geni yigN (nome alternativo sosB), e dinJ (sosA), le caselle SOS non riescono a legare il repressore LexA nonostante i loro valori HI moderati (9.27 e 7.06, rispettivamente) .
Potenziali caselle SOS di geni che legano o non legano il repressore LexA.
| Gene | Sequenza box SOS | HI* | NM** |
|---|---|---|---|
| Consenso | TACTGTATATATACAGTA | 0 | |
| A. Geni le cui scatole SOS legano LexA e sono regolati da LexA represor | |||
| recA | TACTGTATGAGCATACAGTA | 4.31 | 1 |
| umuDC | TACTGTATATAAAAACAGTA | 2.77 | 2 |
| uvrB | AACTGTTTTTATCCAGTA | 6.11 | 5 |
| polB | GACTGTATAAAACCACAGCC | 12.09 | 5 |
| lexA1 | TGCTGTATACTCACAGCA | 6.34 | 4 |
| lexA2 | AACTGTATATACACCCAGGG | 8.32 | 6 |
| B. Geni i cui potenziali SOS box non legano LexA ma non sono regolati da LexA | |||
| intE | GGCTGCTGAAAAATACAGAA | 16.04 | 7 |
| ymfI | TTCTGTACCAGAAAACAGTT | 15.48 | 8 |
| ymfM | AGCTGCAGGAGCATGCAGCA | 19.32 | 3 |
| lit | TGATGACAGAGTGTCCAGTG | 20.32 | 8 |
| C. Geni le cui scatole SOS non legano LexA in spate di basso valore HI | |||
| yigN | AACTGGACGTTTGTACAGCA | 9.27 | 5 |
| dinJ | AGCTGAATAAATATACAGCA | 7.06 | 3 |
Potenziali caselle SOS (sequenza sul filamento codificante) che legano (A), o non legano (B e C) il repressore LexA.
HI* denota l’indice di eterologia; NM** denota il numero di mismatches nelle caselle SOS che deviano da un palindromo perfetto. La mancanza di legame del repressore LexA nonostante un valore di HI relativamente basso (sezione C) testimonia che non c’è una correlazione diretta tra loro. In ogni caso, indica che il valore HI non può essere l’unico indicatore della capacità di un box SOS di legare LexA. Il numero di mismatches nei box SOS palindromici in ciascuna delle sezioni è simile, e quindi non determina la capacità di LexA di legare i box SOS. I dati nelle parti A e C sono da rif.
Caratteristiche di alcuni geni indotti da SOS
I geni di risposta SOS si trovano sparsi nel cromosoma di E. coli come singoli geni situati in singoli operoni. Sei di loro, umuDC (la fonte di Pol V), ruvAB (che catalizza la migrazione dei rami nelle strutture Holliday), e ysdAB (di funzione sconosciuta) sono codificati da coppie di geni che formano un operone. Generalmente, solo una SOS-box è presente in un operone. Le eccezioni sono i geni lexA e ydjM (b1728) che contengono due SOS-box ciascuno (separati da una e due basi, rispettivamente) e recN che contiene tre SOS-box. Le sequenze delle caselle SOS in un gene sono diverse. Nel caso di ydjM, due repressori LexA dimerici si legano in modo cooperativo a ciascuna casella SOS, e come stimato, entrambi sono funzionali. Le sequenze delle SOS-box in un gene differiscono da 2 a 4 basi. Come e perché nascono le SOS-box extra nei geni e come influenzano il potenziale di espressione dei geni sono domande che rimangono senza risposta.
Il tempo richiesto per la derepressione dei geni indotti da SOS
La scala temporale per la derepressione genica e la sintesi delle proteine indotte da SOS varia per i singoli geni. I geni più rapidamente derepressi (<1 min dopo l’induzione SOS) includono: lexA che codifica la proteina repressore LexA (rapidamente degradata nelle cellule indotte da SOS), uvrAB, cho e uvrD coinvolti nella riparazione NER, ruvAB che partecipa alla riparazione ricombinazionale del DNA, polB e dinB che codificano rispettivamente Pol II e Pol IV, e dinI, il cui prodotto inibisce la trasformazione di UmuD in UmuD’ . La proteina UmuD’ è necessaria per la sintesi della Pol V mutagena (UmuD’2C). Pertanto, la proteina DinI ritarda la sintesi di Pol V. L’espressione dei geni recA e recN che codificano per le proteine di ricombinazione e di riparazione ricombinatoria, avviene 5 minuti dopo l’induzione SOS, mentre quella di sulA (vecchio nome, sfiA) e umuDC avviene nell’ultima fase dell’induzione SOS. La proteina sulA è un inibitore della divisione cellulare che causa una crescita filamentosa delle cellule e prolunga il tempo durante il quale il DNA cellulare può essere riparato.
Numeri di copie dei geni din che codificano le proteine
RecA e UvrD appartengono alle proteine più abbondantemente sintetizzate. Il loro numero a livello costitutivo è, rispettivamente, di circa 10.000 e 8.000 copie per cellula e aumenta di 10 volte dopo l’induzione SOS. La proteina RecA si lega al ssDNA e probabilmente lo protegge dalla distruzione incontrollata. UvrD, elicasi II del DNA, partecipa alla riparazione dei mismatch (MMR) mutHLS-dipendente, e prende parte alla riparazione UvrABC- e Cho-dipendente (NER) spostando il ssDNA contenente danni dal filamento di DNA riparato. I numeri di molecole proteiche sintetizzate in cellule non indotte vs. SOS-indotte sono i seguenti: 20:250 per UvrA; 250:1000 per UvrB; 40:300 per DNA Pol II, e 250:2500 per DNA Pol IV (11, 34). La proteina UmuD è espressa, a 180 molecole per non indotto, e a 2400 molecole per cellula repressore LexA mancante funzionale; c’è 200 molecole UmuC per cellula SOS-indotta e nessuna Pol V (< 15 molecole) in cellula non indotta.
Polimerasi del DNA mutagene indotte da SOS
In E. coli, oltre alla Pol III replicante del DNA sintetizzata costitutivamente, ci sono tre polimerasi del DNA potenzialmente mutagene la cui sintesi è aumentata (Pol II e Pol IV) o avviene solo nelle cellule indotte da SOS (Pol V). Tra queste, Pol II è l’unica DNA polimerasi che possiede un’attività di correzione esonucleasica 3′-5′ ed è la meno soggetta a errori; il suo ruolo include anche il recupero del DNA degradato nei forcelle di replicazione. Sia pol II che pol IV appaiono nelle prime fasi dell’induzione SOS, e Pol V nella sua fase finale. Pol V è l’enzima più soggetto a errori e il più importante per la mutagenicità delle cellule indotte da SOS.
In E. coli difettoso in umuD(C) quasi nessuna mutazione è indotta dopo l’irradiazione UV e le mutazioni indotte dal trattamento MMS sono notevolmente ridotte. Le principali lesioni mutagene formate nel DNA dopo irradiazione UV sono TT-cis-syn dimeri ciclobutano e CT o TT (6-4) fotoprodotti , mentre dopo il trattamento MMS, 3-metiladenina (3meA), siti apurinici e 1meA e 3meC (in alkB-mutanti cellule) si trovano predominano. Sia gli UV che gli MMS, come molti altri mutageni, sono induttori di SOS e quindi, anche se le lesioni dannose sono diverse, il segnale che induce SOS deve essere comune; è generalmente accettato che i segnali che inducono SOS sono filamenti di RecA/ssDNA formati su ssDNA accumulato nelle cellule quando la sintesi del DNA viene arrestata. Alcune delle lesioni premutagene richiedono DNA polimerasi mutagene per portare a mutazioni, mentre altre non lo fanno. Tuttavia quale DNA polimerasi mutagena indotta da SOS è richiesta dipende dal tipo di lesione.
Conclusione
L’ipotesi della risposta SOS è stata sorprendente, fruttuosa e stimolante. Abbiamo raccolto molte informazioni sul metabolismo del DNA, l’espressione dei geni indotti da SOS e le loro funzioni. Tuttavia, nuove idee sono ancora in arrivo.
Riconoscimenti
Sono molto grato al professor Graham C. Walker, a Roger Woodgate e alla Blackwell Science Publisher per i permessi di ristampa.
Conflitto di interessi
L’autore ha dichiarato che non esiste alcun conflitto di interessi.
1. Weigle JJ. Induzione di mutazione in un virus batterico. Proc Natl Acad Sci U S A. 1953;39(7):628-636
2. Radman M. Fenomenologia di una via di riparazione del DNA mutagena inducibile in Escherichia coli: ipotesi di riparazione SOS. In: (a cura di) Sherman S, Miller M, Lawrence C, Tabor WH. Aspetti molecolari e ambientali della mutagenesi. Springfield IL: Charles C Thomas editore. 1974:128-142
3. Borek E, Ryan A. Il trasferimento dell’induzione provocata dall’irradiazione in un organismo lisogeno. Proc Natl Acad Sci U S A. 1958;44(5):374-347
4. Hertman I, Luria SE. Studi di trasduzione sul ruolo di un gene rec+ nell’induzione ultravioletta di prophage lambda. J Mol Biol. 1967;23(2):117-133
5. Defais M, Fauquet P, Radman M, Errera M. Riattivazione ultravioletta e mutagenesi ultravioletta di lambda in diversi sistemi genetici. Virologia. 1971;43(2):495-503
6. Craig R. Funzione del trifosfato nucleosidico e del polinucleotide nella scissione diretta dalla proteina recA di Escherichia coli del repressore lambda del fago. J Biol Chem. 1981;256(15):8039-8044
7. Witkin EM. Mutazione indotta dagli ultravioletti e riparazione del DNA. Annu Rev Microbiol. 1969;23:487-514
8. Bridges BA. Riparazione del DNA a rischio di errore e sintesi translesionale del DNA II: L’ipotesi SOS inducibile. Riparazione del DNA (Amst). 2005;4(6):725-739
9. Eller MS, Asarch A, Gilchrest BA. Fotoprotezione nella pelle umana – una risposta SOS multiforme. Phtochem & Photobiol. 2008;84:339-349
10. Clark AJ, Margulies AD. Isolamento e caratterizzazione di mutanti con deficit di ricombinazione di Escherichia coli K12. Proc Natl Acad Sci U S A. 1965;53:451-459
11. Kuzminov A. Riparazione ricombinazionale dei danni al DNA in Escherichia coli e bacteriophage lambda. Microbiol Mol Biol Rev. 1999;63(4):751-813
12. Horii T, Ogawa T, Nakatani T, Hase T, Matsubara H, Ogawa H. Regolazione delle funzioni SOS: purificazione della proteina LexA di E. coli e determinazione del suo sito specifico scisso dalla proteina RecA. Cell. 1981;27(3 Pt 2):515-522
13. Little JW, Mount DW. Il sistema di regolazione SOS di Escherichia coli. Cell. 1982;29(1):11-22
14. Walker GC. Mutagenesi e risposte inducibili al danno dell’acido desossiribonucleico in Escherichia coli. Microbiol Rev. 1984;48(1):60-93
15. Arenson TA, Tsodikov OV, Cox MM. Analisi quantitativa della cinetica del disassemblaggio dipendente dall’estremità dei filamenti RecA da ssDNA. J Mol Biol. 1999;288(3):391-401
16. Schlacher K, Pham P, Cox MM, Goodman MF. Ruoli della DNA polimerasi V e della proteina RecA nella mutazione indotta dal danno SOS. Chem Rev. 2006;106(2):406-419
17. Lewis LK, Harlow GR, Gregg-Jolly LA, Mount DW. Identificazione di siti di legame ad alta affinità per LexA che definiscono nuovi geni inducibili al danno al DNA in Escherichia coli. J Mol Biol. 1994;241(4):507-523
18. Thliveris AT, Little JW, Mount DW. La repressione del gene recA di E coli richiede almeno due monomeri della proteina LexA. Biochimie. 1991;73(4):449-456
19. Burckhardt SE, Woodgate R, Scheuermann RH, Echols H. UmuD mutagenesi proteina di Escherichia coli: sovrapproduzione, purificazione e scissione da RecA. Proc Natl Acad Sci U S A. 1988;85(6):1811-1815
20. Shinagawa H, Iwasaki H, Kato T, Nakata A. RecA protein-dependent cleavage of UmuD protein and SOS mutagenesis. Proc Natl Acad Sci USA. 1988;85(6):1806-1810
21. Tang M, Shen X, Frank EG, O’Donnell M, Woodgate R, Goodman MF. UmuD'(2)C è una DNA polimerasi a rischio di errore, Escherichia coli pol V. Proc Natl Acad Sci U S A. 1999;96(16):8919-8924
22. Kenyon CJ, Walker GC. Gli agenti dannosi per il DNA stimolano l’espressione genica a loci specifici in Escherichia coli. Proc Natl Acad Sci U S A. 1980;77(5):2819-2823
23. Casadaban MJ, Cohen SN. Geni del lattosio fusi a promotori esogeni in un unico passaggio utilizzando un batteriofago Mu-lac: sonda in vivo per sequenze di controllo trascrizionale. Proc Natl Acad Sci U S A. 1979;76(9):4530-4533
24. Bonner CA, Hays S, McEntee K, Goodman MF. La DNA polimerasi II è codificata dal gene dinA, che induce danni al DNA, di Escherichia coli. Proc Natl Acad Sci USA. 1990;87:7663-7667
25. Wagner J, Gruz P, Kim SR, Yamada M, Matsui K, Fuchs RPP, Nohmi T. Il gene dinB codifica una nuova DNA polimerasi di E. coli, DNA pol IV, coinvolta nella mutagenesi. Mol Cell. 1999;4(2):281-286
26. Friedman N, Vardi S, Ronen S, Alin M, Stavans J. Precisa modulazione temporale nella risposta della rete di riparazione del DNA SOS in singoli batteri. PLoS Biol. 2005;3(7):e238
27. Krishna S, Maslov S, Sneppen K. Mutagenesi indotta da UV nella risposta SOS di Escherichia coli: un modello quantitativo. PLoS Comput Biol. 2007;3(3):e41
28. Fernandez de Henestrosa AR, Ogi T, Aoyagi S, Chafin D, Hayes JJ, Ohmori H, Woodgate R. Identificazione di ulteriori geni appartenenti al regulon LexA in Escherichia coli. Mol Microbiol. 2000;35(6):1560-1572
29. Selby CP, Sancar A. Meccanismi di accoppiamento trascrizione-riparazione e declino della frequenza di mutazione. Microbiol Rev. 1994;58(3):317-29
30. Moolenaar GF, van Rossum-Fikkert S, van Kesteren M, Goosen N. Cho, una seconda endonucleasi coinvolta nella riparazione di escissione nucleotidica di Escherichia coli. Proc Natl Acad Sci U S A. 2002;99(3):1467-1472
31. Courcelle J, Khodursky A, Peter B, Brown PO, Hanawalt PC. Profili comparativi di espressione genica dopo l’esposizione ai raggi UV in Escherichia coli wild-type e SOS-deficiente. Genetica. 2001;158(1):41-64
32. Yasuda T, Morimatsu K, Horii T, Nagata T, Ohmori H. Inhibition of Escherichia coli RecA coprotease by Din I. EMBO J. 1998;17(11):3207-3216
33. Cooper DL, Lahue R.S, Modrich P. Mismatch repair in replication fidelity, genetic recombination, and cancer biology. Annu Rev Biochem. 1996;65:101-133
34. Kim S.-R, Matsui K, Yamada M, Gruz P, Nohmi T. Ruoli dei geni din cromosomici che codificano DNA pol IV nella mutagenesi mirata e non mirata in Escherichia coli. Mol Cell. 1999;4(2):281-286
35. Woodgate R, Ennis DG. Livelli di proteine Umu codificate cromosomicamente e requisiti per la scissione UmuD in vivo. Mol Gen Genet. 1991;229(1):10-16
36. Rangarajan S, Woodgate R, Goodman MF. Un fenotipo per l’enigmatica DNA polimerasi II nel riavvio della replicazione in Escherichia coli irradiata dai raggi UV. Proc Natl Acad Sci USA. 1999;96(16):9224-9229
37. Kato T, Shinoura Y. Isolamento e caratterizzazione di mutanti di Escherichia coli carenti nell’induzione di mutazioni da luce ultravioletta. Mol Gen Genet. 1977;156(2):121-131
38. Sledziewska-Gojska E, Janion C. Percorsi alternativi di mutagenesi indotta da metanesulfonato di metile in Escherichia coli. Mol Gen Genet. 1989;216(1):126-31
39. Nieminuszczy J, Sikora A, Wrzesinski M, Janion C, Grzesiuk E. AlkB diossigenasi nella prevenzione della mutagenesi indotta da MMS in Escherichia coli: effetto delle proteine Pol V e AlkA. Riparazione del DNA (Amst). 2006;5(2):181-188
40. Napolitano R, Janel-Bintz R, Wagner J, Fuchs RP. Tutte e tre le DNA polimerasi SOS-inducibili (Pol II, Pol IV e Pol V) sono coinvolte nella mutagenesi indotta. EMBO J. 2000;19(22):6259-6265
41. Fuchs RP, Fujii S, Wagner J. Proprietà e funzioni di Escherichia coli: Pol IV e Pol V. Adv Protein Chem. 2004;69:229-264
42. Foster PL. Mutagenesi indotta da stress nei batteri. Crit Rev Biochem Molec Biol. 2007;42:373-397
43. Iwasaki H, Nakata A, Walker GC, Shinagawa H. Il gene polB di Escherichia coli, che codifica la DNA polimerasi II, è regolato dal sistema SOS. J Bacteriol. 1990;172(11):6268-6273