Int J Biol Sci 2008; 4(6):338-344. doi:10.7150/ijbs.4.338
Review
Celina Janion ![]()
Institut für Biochemie und Biophysik, Polnische Akademie der Wissenschaften, Pawinskiego 5a, 02-106 Warszawa, Polen
Janion C. Inducible SOS Response System of DNA Repair and Mutagenesis in Escherichia coli. Int J Biol Sci 2008; 4(6):338-344. doi:10.7150/ijbs.4.338. Verfügbar unter https://www.ijbs.com/v04p0338.htm
Chromosomale DNA ist ständigen Schäden und Reparaturen ausgesetzt. Die Zellen enthalten eine Reihe von Proteinen und spezifischen DNA-Reparatursystemen, die dazu beitragen, ihre korrekte Struktur zu erhalten. Die SOS-Antwort war das erste DNA-Reparatursystem, das in Escherichia coli beschrieben wurde und durch die Behandlung der Bakterien mit DNA-schädigenden Substanzen ausgelöst wird, wodurch die DNA-Replikation und die Zellteilung unterbrochen werden. An der Induktion der SOS-Reaktion sind mehr als vierzig unabhängige SOS-Gene beteiligt, von denen die meisten für Proteine kodieren, die am Schutz, der Reparatur, der Replikation, der Mutagenese und dem Stoffwechsel der DNA beteiligt sind. Unter normalen Wachstumsbedingungen werden die SOS-Gene auf einem Basalniveau exprimiert, das sich bei Induktion der SOS-Reaktion deutlich erhöht. Die SOS-Antwort wurde in vielen Bakterienarten (z. B. Salmonella typhimurium, Caulobacter crescentus, Mycobacterium tuberculosis), nicht aber in eukaryotischen Zellen gefunden. Arten aus allen Reichen enthalten jedoch einige SOS-ähnliche Proteine, die an der DNA-Reparatur beteiligt sind und eine Aminosäurehomologie und enzymatische Aktivitäten aufweisen, die mit denen in E. coli verwandt sind, aber nicht in einem SOS-System organisiert sind. In diesem Beitrag wird ein kurzer aktueller Überblick über die Entdeckung des SOS-Systems, die Physiologie der SOS-Induktion, Methoden zu seiner Bestimmung und die Rolle einiger SOS-induzierter Gene gegeben.
Schlüsselwörter: SOS-Antwort, DNA-Reparatur, DNA-Mutationen, fehleranfällige Reparatur, mutagene DNA-Polymerasen
Geschichtlicher Überblick
Nachdem erkannt wurde, dass Gene aus DNA bestehen (Oswald T. Avery, 1940), wurden zahlreiche Experimente durchgeführt, um die chemischen Eigenschaften der DNA zu erforschen, vor allem durch die Behandlung von Bakterien und Bakteriophagen mit einer Vielzahl von Agenzien und Chemikalien, wie UV-Licht, Mitomycin C (MC), usw. Auf diese Weise entstand eine wachsende Liste von Bakterienmutanten, die neue und ungewöhnliche Eigenschaften aufwiesen und deren Eigenschaften anschließend bestimmt wurden.
Die Hypothese der SOS-Reaktion wurde auf der Grundlage der folgenden Daten entwickelt: (i) Die Beobachtung von Jean Weigle in l953, dass die Reaktivierung von UV-bestrahlten Phagen λ stark zunahm, wenn die bestrahlten Phagen auf zuvor bestrahlte E. coli-Wirtszellen plattiert wurden. Dieses Phänomen wurde später als W- oder Weigle-Reaktivierung bezeichnet; (ii) Induktion von λ-Prophagen und Lyse von Bakterien (Umwandlung von lysogener zu lytischer Entwicklung) bei UV-Bestrahlung von E. coli-Bakterienlysogenen und (iii) Beobachtung von fadenförmigem Wachstum von E. coli B-Zellen als Reaktion auf UV-Bestrahlung, was auf einen Zusammenhang zwischen dem Stillstand der Zellteilung, den Mechanismen der λ-Prophagen-Induktion und UV-induzierter Mutation schließen lässt. . Diese Daten veranlassten Miroslav Radman zu der Schlussfolgerung, dass es in E. coli ein von den LexA- und RecA-Proteinen abhängiges DNA-Reparatursystem gibt, das induziert wird, wenn die DNA schwer geschädigt und ihre Synthese gestoppt wird, und dass die Induktion dieses Systems mit der Induktion von Mutationen verbunden ist. Radman nannte es „SOS-Reparatur“ und „SOS-Replikation“ nach dem internationalen telegrafischen (oder optischen) Notsignal „SOS“ im Morsealphabet (drei Punkte, drei Striche, drei Punkte).
Die SOS-Hypothese von Miroslav Radman wurde zunächst in einem unveröffentlichten Brief an zahlreiche Forscher im Jahr l970 aufgestellt, der dann erst 1974 veröffentlicht wurde. Evelyn Witkin hatte zuvor die Hypothese aufgestellt, dass die Bildung von Filamenten und die Induktion von Prophagen in bestrahlten E. coli B-Zellen auf einem ähnlichen Mechanismus beruhen könnten. Der ursprüngliche Brief von Radman und die frühe Arbeit von Witkin, die als Grundlage für die Entdeckung des SOS-Reaktionsphänomens gelten, wurden vor kurzem in einer Arbeit von Bryn A. Bridges neu aufgelegt. Weitere Arbeiten in dieser Richtung haben diese Hypothese bestätigt und weiterentwickelt. Später wurde festgestellt, dass Systeme, die in gewisser Hinsicht der in E. coli beschriebenen SOS-Reaktion ähneln, auch in eukaryotischen Zellen funktionieren, aber die bakterielle und die eukaryotische Reaktion unterscheiden sich in der Tat erheblich.
Mechanismus der SOS-Induktion: Rolle der RecA*-Coprotease und des LexA-Repressorproteins
Die recA- und lexA-Gene waren die ersten, die als an der SOS-Induktion beteiligt erkannt wurden. Mutationen in diesen Genen machen Zellen sehr empfindlich gegenüber UV-Bestrahlung. Die 27 kDa LexA- und die 36 kDa RecA-Proteine waren bisher als Rekombinationsproteine bekannt, die am Sexualleben und am genetischen Austausch von Bakterien beteiligt sind. Heute weiß man, dass das RecA-Protein auch am genetischen DNA-Austausch, an der recF-, recO-, recR-, recN- und ruvABC-abhängigen rekombinativen DNA-Reparatur beteiligt ist und zusammen mit dem LexA-Protein eine wichtige Rolle bei der Regulierung der SOS-Reaktion spielt. Die Down- und Up-Regulation der SOS-induzierten Gene ist im Wesentlichen ein Zusammenspiel von zwei Proteinen, LexA-Repressor und RecA*, wobei LexA ein transkriptionelles Repressorprotein und RecA* eine Coprotease ist, die die autokatalytische Selbstspaltung von LexA unterstützt.
Agenten, die das SOS-Reaktionssystem induzieren können, sind z.B., UV-Strahlung, MC, Methylmethansulfonat (MMS) und viele andere Chemikalien, die die DNA zerstören, die DNA-Synthese und die Zellteilung stoppen und zur Anhäufung von einzelsträngiger DNA führen. Die Konzentration des RecA-Proteins in Bakterienzellen ist (wie die der UvrD-Helikase II) sehr hoch. Das RecA-Protein hat eine starke Tendenz zur Bildung von Nukleoproteinfilamenten auf ssDNA, und eine viel schwächere bei gebrochener, doppelsträngiger (ds) DNA. Dies schützt wahrscheinlich die DNA vor Zerstörung und ist für jeden Aspekt der RecA-Aktivität erforderlich. Der Zusammenbau von RecA auf ssDNA erfolgt in 5′-3′-Richtung in einem Verhältnis von 1 Molekül RecA pro 3 DNA-Basen und erfordert dATP oder ATP, aber keine ATP-Aase-Aktivität. Der Abbau hingegen erfordert die Hydrolyse von ATP zu ADP und verläuft viel langsamer als der Zusammenbau. RecA, das an ssDNA assembliert wird, erwirbt eine Coprotease-Aktivität, RecA*, die die Selbstspaltung des LexA-Proteins erleichtert, was zur Derepression von SOS-regulierten Genen führt. Das LexA-Protein hat eine schwache Selbstspaltungsaktivität, aber seine Spaltung und die Derepression der SOS-Gene erfolgen nur in Gegenwart der Coprotease RecA*.
Jedes der SOS-induzierten schadensinduzierbaren (din) oder sos-Gene hat in der Nähe seiner Promotor-/Operatorstelle eine spezifische, 20 Nukleotide lange „SOS-Box“ (auch LexA-Box genannt), an die das LexA-Repressorprotein gebunden ist und die Bindung der RNA-Polymerase und die Genexpression verhindert. Die SOS-Box hat eine palindromische Struktur, was darauf hindeutet, dass der LexA-Repressor als Dimer bindet, wie später bestätigt wurde. Die Rolle der RecA*-Coprotease in SOS-induzierten Zellen besteht also darin: 1. die Spaltung des LexA-Proteins (202 Aminosäuren) an der Ala84-Gly85-Stelle zu unterstützen, was die Derepression der SOS-Gene bewirkt; 2. Spaltung des CI-Repressors des λ-Lambda-Phagen, wodurch der Phage von einer lysogenen in eine lytische Form umgewandelt wird; 3. Verarbeitung von UmuD → UmuD‘ durch Nicking von UmuD an der Cys24-Gly25-Stelle, was eine Voraussetzung für den Aufbau der SOS-induzierten mutagenen DNA-Polymerase V (Pol V) ist, die aus UmuD’2C besteht. Der geschwindigkeitsbegrenzende Schritt der Pol V-Synthese ist die UmuD→ UmuD‘-Prozessierung, die viel langsamer abläuft als die Selbstspaltung von LexA. Die Rolle von Pol V bei der Mutagenese besteht in der Translesionssynthese (TLS) über den Schaden in der Template-DNA, wodurch die DNA-Replikation ermöglicht wird, häufig auf Kosten der Treue, was zu Mutationen führt. Alle diese Proteine, der CI-Repressor des λ-Phagen und der LexA-Repressor, UmuD, PolB/DinA (Pol II) und DinB (Pol IV) sind in ihren carboxyterminalen Domänen homolog, und alle werden von Din (sos)-Genen kodiert, die im Rahmen der SOS-Antwort reguliert werden.
Die Induktion der SOS-Antwort dauert bis zu 45-60 Minuten nach der Behandlung der Bakterien mit SOS-induzierenden Agenzien und hört dann abrupt auf. Innerhalb dieses Zeitraums sind die meisten Läsionen repariert worden. Der Zeitpunkt der Derepression einzelner din-Gene hängt von der Stärke der Bindung des LexA-Repressors an die SOS-Box und von der Leichtigkeit ab, mit der der LexA-Repressor von einer bestimmten SOS-Box abgelöst wird.
3.1. Durch din::lacZ-Bildung und β-Galaktosidase-Assay
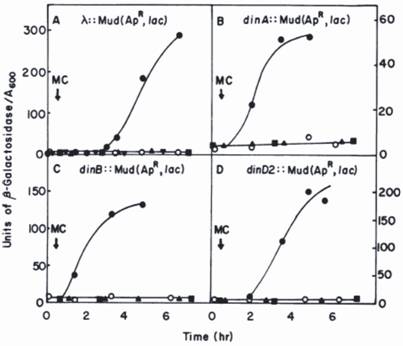
Die SOS-Antwort wurde früher untersucht, indem man die Zunahme der din-Genexpression entweder aus den natürlichen Genen oder mit Hilfe eines Reportergenkonstrukts, z.B. der Fusion eines putativen din-Promotors mit dem promotorlosen lacZ-Gen, das für β-Galaktosidase kodiert, testete. Graham Walker und seine Mitarbeiter waren die ersten, die für diese Aufgabe einen defekten Phagen, Mu1d(Ap,lacZ), konstruiert von Casadaban und Cohen, einsetzten, der sich leicht zufällig in das Chromosom von E. coli K12 einfügt und eine Mutation erzeugt. Dieser Phage trägt ein promotorloses lacZ-Gen, so dass die β-Galaktosidase nicht exprimiert wird. Wenn der Mu-Phagen jedoch zufällig unter dem Promotor eines din-Gens integriert wird und ein funktionelles din::Mu-1d(Ap,lacZ)-Operon bildet, wird als Reaktion auf DNA-Schäden β-Galaktosidase synthetisiert.
Da die Hydrolyse des β-Galaktosidase-Substrats (o-Nitrophenyl-β-galaktosid) gelbe Kolonien bildet, können diejenigen, die eine din::Mu-lacZ-Fusion tragen, leicht auf Agarplatten selektiert werden; der genaue Gehalt an β-Galaktosidase, die als Reaktion auf DNA-Schäden exprimiert wird, kann dann in flüssigem Medium genau gemessen werden (siehe Abb.1 für Details). Auf diese Weise wurden mehr als zehn neue din-Gene entdeckt und die meisten von ihnen anschließend identifiziert.
Kinetik der Induktion von β-Galactosidase in din::lacZ-Fusionsstämmen durch Mitomycin C (MC) . Die din::lac-Fusionen wurden durch die Insertion des Mu d1(Ap, lac)-Bakteriophagen in das E. coli-Chromosom erzeugt. Das λ::Mu d1(Ap lac)-Derivat wurde durch Insertion von Mu d1(Ap lac) in den λ-Phagen in das E. coli-Chromosom erzeugt. Symbole: o, unbehandelter Fusionsstamm; ●, Fusionsstämme plus MC; lexA(Ind-) Derivate des Fusionsstammes plus MC; ■, recA (Def) Derivate des Fusionsstammes plus MC; ▼, ein pKM280-haltiges Derivat des λ:: Mu d1(Ap lac)-Stammes plus MC. Nachdruck mit Genehmigung des Autors. Zwei der Gene, dinA und dinB, wurden später als polB (Pol II) bzw. Pol IV identifiziert.
 (Klicken Sie auf das Bild, um es zu vergrößern.)
(Klicken Sie auf das Bild, um es zu vergrößern.)
In jüngster Zeit wurde eine neue Methode entwickelt, um die SOS-Genexpression und die Promotoraktivität der SOS-Gene (z.B., recA, lexA, umuDC) zu messen, indem ein Plasmid verwendet wird, das einen zu untersuchenden SOS-Promotor trägt, der mit dem Reportergen gfp, das für grün fluoreszierendes Protein (GFP) kodiert, fusioniert ist. Auf diese Weise kann die Promotoraktivität von SOS-Genen in einer einzigen Bakterienzelle gemessen werden, ebenso wie die Lokalisierung und Dauer der SOS-Induktion. Es zeigt sich, dass die Induktion der SOS-Gene nicht in einem einzigen Vorgang abläuft, sondern in mehreren wiederholbaren Schritten, deren Modulation von der SOS-induzierenden Dosis, dem Grad der Schädigung der DNA und der Akkumulation des UmuD‘-Proteins abhängt. Diese Methode eröffnet einen neuen Weg zur Messung der Dynamik der SOS-Reaktion.
3.2. Durch die Suche nach SOS-Boxen: Bestimmung des Heterologie-Index (HI)
Fortschritte in der DNA-Sequenzierung und die Kenntnis der charakteristischen Elemente von SOS-Gensequenzen ermöglichten eine direkte rechnerische Suche nach SOS-induzierbaren Genen. Nachdem 33 % der chromosomalen DNA von E. coli sequenziert worden waren, lokalisierten Lewis et al. durch Sequenzanalyse und quantitative DNA-Bindungsexperimente sechs neue, potenziell LexA-regulierte Gene und nannten sie sosA-F . Für zwei von ihnen, sosC und sosD, bestätigten die Autoren experimentell, dass sie gereinigten LexA-Repressor stark binden.
Durch den Vergleich der Sequenzen der SOS-Boxen von 19 damals bekannten Din-Genen (einschließlich sosA-sosF) stellten sie fest, dass die Konsenssequenz der SOS-Boxen ein perfektes Palindrom ist, TACTG(TA)5CAGTA ; und auf der Grundlage der Theorie von Berg und von Hippel berechneten sie mathematisch für jede der SOS-Boxen einen Heterologieindex (HI). Dieser Index zeigt die Abweichung einer SOS-Box vom Konsens an, und wenn sein Wert niedrig ist, wird das Gen stark unterdrückt, und wenn sein Wert hoch ist, wird es leichter de-reprimiert. Bei einem HI-Wert von mehr als 15 bindet der LexA-Repressor nicht an die SOS-Box. Der HI-Wert ist somit ein Maß für die relative Stärke der Bindung des LexA-Repressors an eine bestimmte SOS-Box und ist für die Variation des Depressionspotenzials verantwortlich.
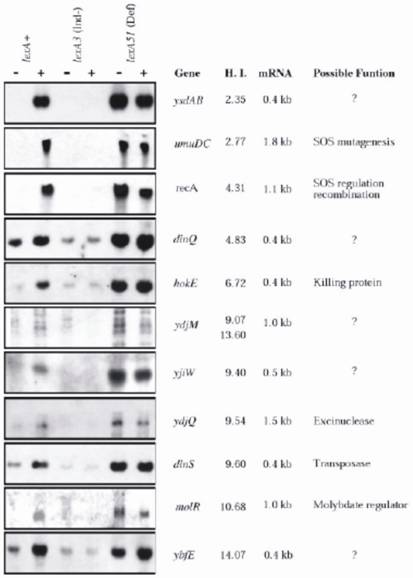
Nachdem die DNA-Sequenz des gesamten E. coli K12-Chromosoms bestimmt worden war, lokalisierten Fernandez de Henestrosa et al. durch die Suche nach potenziellen SOS-Boxen, die mit offenen Leserahmen assoziiert sind, 69 potenzielle SOS-Boxen mit einem HI-Wert ≤ 15, darunter alle bisher bekannten und sieben neue. Die neuen Gene wurden anschließend auf ihre Fähigkeit, bei MC-Behandlung exprimiert zu werden, auf die Länge der exprimierten mRNA und auf die HI-Werte untersucht (Abb. 2). Die Analysen wurden in drei isogenen E. coli-Stämmen durchgeführt, die sich durch das lexA-Allel unterscheiden: lexA+(Wildtyp) SOS-induzierbar, SOS nicht-induzierbar lexA3(Ind-) und das konstitutiv exprimierte lexA51(Def)-Allel. Die möglichen Funktionen der neuen und alten Gene wurden weiter charakterisiert und diskutiert. Die Ergebnisse bestätigten, dass jedes der neuen SOS-Box-enthaltenden Gene in der Tat ein LexA-abhängiges Gen war und seine Expression durch MC nur im lexA+-Stamm induziert wurde; ansonsten wurden sie entweder nicht exprimiert (lexA3) oder vollständig exprimiert (lexA51), unabhängig davon, ob die Bakterien mit MC behandelt wurden oder nicht (siehe Abb. 2 für Details).
Aus der Nukleotidsequenz des ydjQ-Gens (alternative Namen b1741 sosD) wurde abgeleitet, dass es für ein 295 Aminosäuren langes Protein kodiert, das eine signifikante Homologie mit der N-terminalen Hälfte des UvrC-Proteins aufweist. Es war bekannt, dass die UvrABC-Excinuklease (bestehend aus den Proteinen UvrA, UvrB und UvrC) an der Nukleotidexzisionsreparatur (NER) beteiligt ist, die sperrige Addukte oder strukturbeeinträchtigende Läsionen (z. B. Pyrimidindimere, Pyrimidin-(6-4)-Photoprodukte und abasische Stellen) aus modifizierter DNA entfernt. Es war auch bekannt, dass der N-Teil von UvrC die ssDNA zunächst an der 3′-Seite der Läsion einschneidet, und dann der C-Teil von UvrC die DNA an der 5′-Seite der Läsion einschneidet. Kürzlich haben Moolenaar et al. das YdjQ-Protein in Cho umbenannt (nach dem UvrC-Homolog) und seine enzymatische Aktivität untersucht. Sie bestätigten, dass das Cho-Protein die UvrB-DNA-Vorschneidekomplexe nur an der 3′-Seite der Läsion einschneidet; einige Läsionen in der DNA, die vom UvrC-Protein sehr schlecht eingeschnitten wurden, wurden jedoch vom Cho-Protein sehr effizient eingeschnitten. Das Cho-Protein vergrößert also den Substratbereich bei der DNA-Reparatur durch das NER-System erheblich. Die uvrA-, uvrB- und ydjQ-Gene (aber nicht uvrC) sind SOS-induzierte Gene.
Northern-Analyse von E. coli-Genen, die offenbar durch LexA reguliert werden. Die RNA wurde aus drei isogenen Stämmen extrahiert, die sich im LexA-Gen unterscheiden: RW118 (lexA+), RW434 (lexA) und RW542 (lexA51). Die RNA wurde aus unbeschädigten Zellen (-) und aus Zellen, die vor der Extraktion 30 Minuten lang mit Mitomycin C (5 µg ml-1) (+) behandelt worden waren, gewonnen. Die zuvor identifizierten LexA-regulierten recA- und umuDC-Gene wurden als Positivkontrollen verwendet. Die Gene sind entsprechend ihrem aufsteigenden Heterologieindex (HI) dargestellt. Die Größe der mRNA-Transkripte und die möglichen Funktionen der Gene sind ebenfalls angegeben Weitere Einzelheiten finden Sie unter Ref. (Mit freundlicher Genehmigung von Blackwell Science).
 (Zum Vergrößern auf das Bild klicken.)
(Zum Vergrößern auf das Bild klicken.)
3.3. Lokalisierung von din-Genen durch Microarray-Techniken
Die Microarray-Technik ermöglicht es, eine große Anzahl von Genen in einem Experiment zu überwachen. Courcelle et al. verwendeten Microarrays, die amplifizierte E coli DNA-Chromosomenfragmente mit offenen Leserahmen von 4101 Genen (95,5 % der Gesamtmenge) enthielten, um die Expression aller Gene in UV-bestrahlten und nicht-bestrahlten SOS-induzierbaren (lexA+) und nicht-induzierbaren lexA1(Ind-)Stämmen zu messen. In dem UV-bestrahlten lexA+-Stamm identifizierten die Autoren 17 neue LexA-abhängige SOS-induzierte Gene, zusätzlich zu den 26 bereits bekannten; die Gesamtzahl der SOS-induzierbaren Gene in E. coli beträgt daher wahrscheinlich 43. In derselben Publikation stellten die Autoren fest, dass das ssb-Gen, das für ein ssDNA-bindendes Protein kodiert, nicht SOS-induzierbar ist, wie bisher angenommen wurde. Sie beobachteten auch eine Reihe von Genen, deren Expression in UV-bestrahlten Zellen anstieg (in der Regel nicht über das Zweifache), die aber nicht durch das LexA-Protein reguliert wurden. Sie stellten fest, dass die Proteintranskripte vieler Gene, die nicht durch LexA reguliert werden, nach der UV-Bestrahlung reduziert waren, und schlossen daraus, dass diese Transkripte wahrscheinlich durch die UV-Strahlung entweder beschädigt oder abgebaut wurden. Sie identifizierten auch dreißig Gene mit potenziellen SOS-Box-ähnlichen Strukturen, die jedoch nicht von LexA reguliert wurden.
Mechanismus und Spezifität der Bindung des LexA-Repressors an SOS-Boxen
Es wird angenommen, dass die Sequenzen aller potenziellen SOS-Boxen im E. coli-Chromosom identifiziert worden sind. Einige Beispiele für SOS-Boxen, die den LexA-Repressor binden (A) oder nicht binden (B und C), sind zusammen mit den HI-Werten und der Anzahl der Fehlpaarungen (NM) in Tabelle 1 aufgeführt. NM bezeichnet die Anzahl der Positionen in den SOS-Boxen, die von einem perfekten Palindrom abweichen. Sowohl die Anzahl als auch das Muster der Fehlanpassungen können entscheidend für die Spezifität der Bindung des LexA-Proteins an jede einzelne SOS-Box sein. Es scheint, dass diese Hypothese eine gute Erklärung für die Spezifität und die unterschiedliche Stärke der Bindung des LexA-Proteins an die Sequenzen der SOS-Boxen ist. Dies sollte jedoch bestätigt werden.
Es zeigt sich, dass die SOS-Boxen mit niedrigen HI-Werten zwischen 2,7 und 12 im Allgemeinen den LexA-Repressor binden können (Tabelle 1A); liegt der HI-Wert über 16,4 (Tabelle 1B), sind sie offenbar nicht in der Lage, den LexA-Repressor zu binden. In einigen Fällen (siehe Teil C), wie z. B. bei den Genen yigN (alternativer Name sosB) und dinJ (sosA), gelingt es den SOS-Boxen jedoch nicht, den LexA-Repressor zu binden, trotz ihrer moderaten HI-Werte (9,27 bzw. 7,06).
Potenzielle SOS-Boxen von Genen, die den LexA-Repressor binden oder nicht binden.
| Gen | SOS-Box-Sequenz | HI* | NM** |
|---|---|---|---|
| Konsens | TACTGTATATATATATACAGTA | 0 | |
| A. Gene, deren SOS-Boxen LexA binden und durch LexA represor | |||
| recA | TACTGTATGAGCATACAGTA | 4 reguliert werden.31 | 1 |
| umuDC | TACTGTATATAAAAACAGTA | 2.77 | 2 |
| uvrB | AACTGTTTTTTATCCAGTA | 6.11 | 5 |
| polB | GACTGTATAAAACCACAGCC | 12.09 | 5 |
| lexA1 | TGCTGTATATACTCACAGCA | 6.34 | 4 |
| lexA2 | AACTGTATATACACCCAGGG | 8.32 | 6 |
| B. Gene, deren potenzielle SOS-Boxen LexA nicht binden, aber nicht LexA-reguliert sind | |||
| intE | GGCTGCTGAAAAATACAGAA | 16.04 | 7 |
| ymfI | TTCTGTACCAGAAAACAGTT | 15.48 | 8 |
| ymfM | AGCTGCAGGAGCATGCAGCA | 19.32 | 3 |
| lit | TGATGACAGAGTGTCCAGTG | 20.32 | 8 |
| C. Gene, deren SOS-Boxen LexA nicht binden, haben einen niedrigen HI-Wert | |||
| yigN | AACTGGACGTTTGTACAGCA | 9.27 | 5 |
| dinJ | AGCTGAATAAATATACAGCA | 7.06 | 3 |
Potenzielle SOS-Boxen (Sequenz auf dem kodierenden Strang), die den LexA-Repressor binden (A) oder nicht binden (B und C).
HI* bezeichnet den Heterologie-Index; NM** bezeichnet die Anzahl der Fehlpaarungen in SOS-Boxen, die von einem perfekten Palindrom abweichen. Die fehlende Bindung des LexA-Repressors trotz eines relativ niedrigen HI-Wertes (Abschnitt C) beweist, dass es keine direkte Korrelation zwischen ihnen gibt. Es zeigt jedoch, dass der HI-Wert nicht der einzige Indikator für die Fähigkeit einer SOS-Box sein kann, LexA zu binden. Die Anzahl der Mismatches in den palindromischen SOS-Boxen in jedem der Abschnitte ist ähnlich und bestimmt daher nicht die Fähigkeit von LexA, an die SOS-Boxen zu binden. Die Daten in den Teilen A und C stammen aus ref.
Charakteristika einiger SOS-induzierter Gene
Die SOS-Antwortgene sind über das gesamte E. coli-Chromosom verstreut als Einzelgene in einzelnen Operons zu finden. Sechs von ihnen, umuDC (die Quelle von Pol V), ruvAB (katalysiert die Verzweigungswanderung in Holliday-Strukturen) und ysdAB (mit unbekannter Funktion) werden von Genpaaren kodiert, die ein Operon bilden. Im Allgemeinen ist nur eine SOS-Box in einem Operon vorhanden. Ausnahmen sind die Gene lexA und ydjM (b1728), die jeweils zwei SOS-Boxen enthalten (getrennt durch eine bzw. zwei Basen), und recN mit drei SOS-Boxen. Die Sequenzen der SOS-Boxen in einem Gen sind unterschiedlich. Im Fall von ydjM binden zwei dimere LexA-Repressoren kooperativ an jede SOS-Box, und wie geschätzt, sind beide funktionell. Die Sequenzen der SOS-Boxen in einem Gen unterscheiden sich um 2 bis 4 Basen. Wie und warum die zusätzlichen SOS-Boxen in Genen entstehen und wie sie das Genexpressionspotenzial beeinflussen, sind Fragen, die noch zu beantworten sind.
Die für die Derepression von SOS-induzierten Genen erforderliche Zeit
Die Zeitskala für die Gendepression und die Synthese der SOS-induzierten Proteine variiert für einzelne Gene. Zu den am schnellsten dereprimierten Genen (<1 min nach SOS-Induktion) gehören: lexA, das für das LexA-Repressorprotein kodiert (das in SOS-induzierten Zellen schnell abgebaut wird), uvrAB, cho und uvrD, die an der NER-Reparatur beteiligt sind, ruvAB, das an der rekombinativen DNA-Reparatur teilnimmt, polB und dinB, die für Pol II bzw. Pol IV kodieren, und dinI, dessen Produkt die Prozessierung von UmuD zu UmuD‘ verhindert. Das UmuD‘-Protein ist für die Synthese des mutagenen Pol V (UmuD’2C) notwendig. Daher verzögert das DinI-Protein die Synthese von Pol V. Die Expression der recA- und recN-Gene, die für Rekombinations- und Rekombinationsreparaturproteine kodieren, erfolgt 5 Minuten nach der SOS-Induktion, während die Expression von sulA (alter Name: sfiA) und umuDC in der letzten Phase der SOS-Induktion erfolgt. Das SulA-Protein ist ein Inhibitor der Zellteilung, der ein fadenförmiges Wachstum der Zellen verursacht und die Zeit verlängert, in der die zelluläre DNA repariert werden kann.
Kopienzahlen von din Genen, die Proteine kodieren
RecA und UvrD gehören zu den am häufigsten synthetisierten Proteinen. Ihre Anzahl beträgt konstitutiv etwa 10.000 bzw. 8.000 Kopien pro Zelle und steigt nach SOS-Induktion um das 10-fache. Das RecA-Protein bindet an ssDNA und schützt sie wahrscheinlich vor unkontrollierter Zerstörung. UvrD, die DNA-Helikase II, ist an der Dam-instruierten mutHLS-abhängigen Mismatch-Reparatur (MMR) beteiligt und nimmt an der UvrABC- und Cho-abhängigen (NER) Reparatur teil, indem es die schadensbehaftete ssDNA vom reparierten DNA-Strang verdrängt. Die Anzahl der Proteinmoleküle, die in nicht induzierten Zellen im Vergleich zu SOS-induzierten Zellen synthetisiert werden, ist wie folgt: 20:250 für UvrA; 250:1000 für UvrB; 40:300 für DNA Pol II, und 250:2500 für DNA Pol IV (11, 34). Das UmuD-Protein wird exprimiert, und zwar 180 Moleküle pro nicht induzierter und 2400 Moleküle pro nicht funktionsfähiger LexA-Repressorzelle; es gibt 200 UmuC-Moleküle pro SOS-induzierter Zelle und kein Pol V (< 15 Moleküle) in nicht induzierter Zelle.
Mutagene SOS-induzierte DNA-Polymerasen
In E. coli gibt es neben der konstitutiv synthetisierten DNA-replizierenden Pol III drei potentiell mutagene DNA-Polymerasen, deren Synthese erhöht ist (Pol II und Pol IV) oder nur in SOS-induzierten Zellen auftritt (Pol V). Unter diesen ist Pol II die einzige DNA-Polymerase, die eine 3′-5′-Exonuklease-Korrekturleseaktivität besitzt und die am wenigsten fehleranfällig ist; zu ihren Aufgaben gehört auch die Wiederherstellung von abgebauter DNA an Replikationsgabeln. Sowohl Pol II als auch Pol IV treten in den frühen Phasen der SOS-Induktion auf, Pol V in der Endphase. Pol V ist das fehleranfälligste Enzym und das wichtigste für die Mutagenität der SOS-induzierten Zellen.
In E. coli mit Defekt in umuD(C) werden nach UV-Bestrahlung fast keine Mutationen induziert und die durch MMS-Behandlung induzierten Mutationen sind stark reduziert. Die wichtigsten mutagenen Läsionen, die nach UV-Bestrahlung in der DNA gebildet werden, sind TT-cis-syn-Cyclobutan-Dimere und CT- oder TT-(6-4)-Photoprodukte, während nach MMS-Behandlung vor allem 3-Methyladenin (3meA), apurinische Stellen sowie 1meA und 3meC (in alkB-mutanten Zellen) gefunden werden. Sowohl UV als auch MMS sind, wie viele andere Mutagene, SOS-Induktoren, und obwohl die schädigenden Läsionen unterschiedlich sind, muss das SOS-induzierende Signal ein gemeinsames sein; es wird allgemein angenommen, dass SOS-induzierende Signale RecA/ssDNA-Filamente sind, die sich bei der Anhäufung von ssDNA in den Zellen bilden, wenn die DNA-Synthese gestoppt ist. Einige der prämutagenen Läsionen erfordern mutagene DNA-Polymerasen, um zu Mutationen zu führen, andere dagegen nicht. Welche SOS-induzierte, mutagene DNA-Polymerase jedoch erforderlich ist, hängt von der Art der Läsion ab.
Schlussfolgerung
Die Hypothese der SOS-Reaktion war erstaunlich, fruchtbar und inspirierend. Wir haben viele Informationen über den Stoffwechsel der DNA, die Expression der SOS-induzierten Gene und ihre Funktionen gesammelt. Dennoch gibt es immer noch neue Ideen.
Danksagung
Ich bin Professor Graham C. Walker, Roger Woodgate und dem Blackwell Science Verlag sehr dankbar für die Abdruckgenehmigung.
Interessenkonflikt
Der Autor hat erklärt, dass kein Interessenkonflikt besteht.
1. Weigle JJ. Induktion von Mutation in einem bakteriellen Virus. Proc Natl Acad Sci U S A. 1953;39(7):628-636
2. Radman M. Phänomenologie eines induzierbaren mutagenen DNA-Reparaturweges in Escherichia coli: SOS-Reparaturhypothese. In: (ed.) Sherman S, Miller M, Lawrence C, Tabor WH. Molekulare und umweltbezogene Aspekte der Mutagenese. Springfield IL: Charles C Thomas publisher. 1974:128-142
3. Borek E, Ryan A. The transfer of irradiation-elicited induction in a lysogenic organism. Proc Natl Acad Sci U S A. 1958;44(5):374-347
4. Hertman I, Luria SE. Transduction studies on the role of a rec+ gene in the ultraviolet induction of prophage lambda. J Mol Biol. 1967;23(2):117-133
5. Defais M, Fauquet P, Radman M, Errera M. Ultraviolet reactivation and ultraviolet mutagenesis of lambda in different genetic systems. Virology. 1971;43(2):495-503
6. Craig R. Function of nucleoside triphosphate and polynucleotide in Escherichia coli recA protein-directed cleavage of phage lambda repressor. J Biol Chem. 1981;256(15):8039-8044
7. Witkin EM. Ultraviolett-induzierte Mutation und DNA-Reparatur. Annu Rev Microbiol. 1969;23:487-514
8. Bridges BA. Fehleranfällige DNA-Reparatur und Translesion-DNA-Synthese II: Die induzierbare SOS-Hypothese. DNA Repair (Amst). 2005;4(6):725-739
9. Eller MS, Asarch A, Gilchrest BA. Photoprotection in human skin-A multifaceted SOS response. Phtochem & Photobiol. 2008;84:339-349
10. Clark AJ, Margulies AD. Isolation und Charakterisierung von rekombinationsdefizienten Mutanten von Escherichia coli K12. Proc Natl Acad Sci U S A. 1965;53:451-459
11. Kuzminov A. Recombinational repair of DNA damage in Escherichia coli and bacteriophage lambda. Microbiol Mol Biol Rev. 1999;63(4):751-813
12. Horii T, Ogawa T, Nakatani T, Hase T, Matsubara H, Ogawa H. Regulation of SOS functions: purification of E. coli LexA protein and determination of its specific site cleaved by the RecA protein. Cell. 1981;27(3 Pt 2):515-522
13. Little JW, Mount DW. Das SOS-Regulationssystem von Escherichia coli. Cell. 1982;29(1):11-22
14. Walker GC. Mutagenese und induzierbare Reaktionen auf Desoxyribonukleinsäure-Schäden in Escherichia coli. Microbiol Rev. 1984;48(1):60-93
15. Arenson TA, Tsodikov OV, Cox MM. Quantitative Analyse der Kinetik des endabhängigen Abbaus von RecA-Filamenten aus ssDNA. J Mol Biol. 1999;288(3):391-401
16. Schlacher K, Pham P, Cox MM, Goodman MF. Roles of DNA polymerase V and RecA protein in SOS damage-induced mutation. Chem Rev. 2006;106(2):406-419
17. Lewis LK, Harlow GR, Gregg-Jolly LA, Mount DW. Identifizierung von hochaffinen Bindungsstellen für LexA, die neue DNA-Schaden-induzierbare Gene in Escherichia coli definieren. J Mol Biol. 1994;241(4):507-523
18. Thliveris AT, Little JW, Mount DW. Die Repression des E coli recA-Gens erfordert mindestens zwei LexA-Proteinmonomere. Biochimie. 1991;73(4):449-456
19. Burckhardt SE, Woodgate R, Scheuermann RH, Echols H. UmuD mutagenesis protein of Escherichia coli: overproduction, purification, and cleavage by RecA. Proc Natl Acad Sci U S A. 1988;85(6):1811-1815
20. Shinagawa H, Iwasaki H, Kato T, Nakata A. RecA protein-dependent cleavage of UmuD protein and SOS mutagenesis. Proc Natl Acad Sci USA. 1988;85(6):1806-1810
21. Tang M, Shen X, Frank EG, O’Donnell M, Woodgate R, Goodman MF. UmuD'(2)C ist eine fehleranfällige DNA-Polymerase, Escherichia coli pol V. Proc Natl Acad Sci U S A. 1999;96(16):8919-8924
22. Kenyon CJ, Walker GC. DNA-schädigende Agenzien stimulieren die Genexpression an spezifischen Loci in Escherichia coli. Proc Natl Acad Sci U S A. 1980;77(5):2819-2823
23. Casadaban MJ, Cohen SN. Lactose Gene fusioniert mit exogenen Promotoren in einem Schritt mit einem Mu-lac Bakteriophagen: in vivo Sonde für transkriptionelle Kontrollsequenzen. Proc Natl Acad Sci U S A. 1979;76(9):4530-4533
24. Bonner CA, Hays S, McEntee K, Goodman MF. DNA Polymerase II wird durch das DNA-Schaden-induzierbare dinA-Gen von Escherichia coli kodiert. Proc Natl Acad Sci USA. 1990;87:7663-7667
25. Wagner J, Gruz P, Kim SR, Yamada M, Matsui K, Fuchs RPP, Nohmi T. The dinB gene encodes a novel E. coli DNA polymerase, DNA pol IV, involved in mutagenesis. Mol Cell. 1999;4(2):281-286
26. Friedman N, Vardi S, Ronen S, Alin M, Stavans J. Precise temporal modulation in the response of the SOS DNA repair network in individual bacteria. PLoS Biol. 2005;3(7):e238
27. Krishna S, Maslov S, Sneppen K. UV-induced mutagenesis in Escherichia coli SOS response: a quantitative model. PLoS Comput Biol. 2007;3(3):e41
28. Fernandez de Henestrosa AR, Ogi T, Aoyagi S, Chafin D, Hayes JJ, Ohmori H, Woodgate R. Identification of additional genes belonging to the LexA regulon in Escherichia coli. Mol Microbiol. 2000;35(6):1560-1572
29. Selby CP, Sancar A. Mechanisms of transcription-repair coupling and mutation frequency decline. Microbiol Rev. 1994;58(3):317-29
30. Moolenaar GF, van Rossum-Fikkert S, van Kesteren M, Goosen N. Cho, a second endonuclease involved in Escherichia coli nucleotide excision repair. Proc Natl Acad Sci U S A. 2002;99(3):1467-1472
31. Courcelle J, Khodursky A, Peter B, Brown PO, Hanawalt PC. Vergleichende Genexpressionsprofile nach UV-Exposition in Wildtyp und SOS-defizienten Escherichia coli. Genetics. 2001;158(1):41-64
32. Yasuda T, Morimatsu K, Horii T, Nagata T, Ohmori H. Inhibition of Escherichia coli RecA coprotease by Din I. EMBO J. 1998;17(11):3207-3216
33. Cooper DL, Lahue R.S, Modrich P. Mismatch repair in replication fidelity, genetic recombination, and cancer biology. Annu Rev Biochem. 1996;65:101-133
34. Kim S.-R, Matsui K, Yamada M, Gruz P, Nohmi T. Roles of chromosomal din genes encoding DNA pol IV in targeted and untargeted mutagenesis in Escherichia coli. Mol Cell. 1999;4(2):281-286
35. Woodgate R, Ennis DG. Levels of chromosomally encoded Umu proteins and requirements for in vivo UmuD cleavage. Mol Gen Genet. 1991;229(1):10-16
36. Rangarajan S, Woodgate R, Goodman MF. Ein Phänotyp für die rätselhafte DNA-Polymerase II beim Replikationsneustart in UV-bestrahlten Escherichia coli. Proc Natl Acad Sci USA. 1999;96(16):9224-9229
37. Kato T, Shinoura Y. Isolation and characterization of mutants of Escherichia coli deficient in induction of mutations by ultraviolet light. Mol Gen Genet. 1977;156(2):121-131
38. Sledziewska-Gojska E, Janion C. Alternative Wege der Methylmethansulfonat-induzierten Mutagenese in Escherichia coli. Mol Gen Genet. 1989;216(1):126-31
39. Nieminuszczy J, Sikora A, Wrzesinski M, Janion C, Grzesiuk E. AlkB dioxygenase in preventing MMS-induced mutagenesis in Escherichia coli: effect of Pol V and AlkA proteins. DNA Repair (Amst). 2006;5(2):181-188
40. Napolitano R, Janel-Bintz R, Wagner J, Fuchs RP. Alle drei SOS-induzierbaren DNA-Polymerasen (Pol II, Pol IV und Pol V) sind an der induzierten Mutagenese beteiligt. EMBO J. 2000;19(22):6259-6265
41. Fuchs RP, Fujii S, Wagner J. Properties and functions of Escherichia coli: Pol IV and Pol V. Adv Protein Chem. 2004;69:229-264
42. Foster PL. Stress-induzierte Mutagenese in Bakterien. Crit Rev Biochem Molec Biol. 2007;42:373-397
43. Iwasaki H, Nakata A, Walker GC, Shinagawa H. The Escherichia coli polB gene, which encodes DNA polymerase II, is regulated by the SOS system. J Bacteriol. 1990;172(11):6268-6273