Int J Biol Sci 2008; 4(6):338-344. doi:10.7150/ijbs.4.338
Revisão
Celina Janion ![]()
Instituto de Bioquímica e Biofísica, Academia Polonesa de Ciências, Pawinskiego 5a, 02-106 Warszawa, Polônia
Janion C. Sistema Inducivel de Resposta SOS de Reparação e Mutagénese de DNA em Escherichia coli. Int J Biol Sci 2008; 4(6):338-344. doi:10.7150/ijbs.4.338. Disponível a partir de https://www.ijbs.com/v04p0338.htm
O DNA cromossómico está exposto a danos e reparações contínuas. As células contêm uma série de proteínas e sistemas específicos de reparação do ADN que ajudam a manter a sua estrutura correcta. A resposta SOS foi o primeiro sistema de reparação de ADN descrito na Escherichia coli induzido no tratamento de bactérias com agentes danificadores do ADN que impedem a replicação do ADN e a divisão celular. A indução da resposta SOS envolve mais de quarenta genes SOS independentes, a maioria dos quais codificam proteínas envolvidas na proteção, reparação, replicação, mutagênese e metabolismo do DNA. Sob condições normais de crescimento os genes SOS são expressos a um nível basal, que aumenta distintamente após a indução da resposta SOS. A resposta SOS tem sido encontrada em muitas espécies bacterianas (por exemplo, Salmonella typhimurium, Caulobacter crescentus, Mycobacterium tuberculosis), mas não em células eucarióticas. Entretanto, espécies de todos os reinos contêm algumas proteínas do tipo SOS que participam do reparo do DNA que exibem homologia de aminoácidos e atividades enzimáticas relacionadas àquelas encontradas na E. coli. mas não são organizadas em um sistema SOS. Este artigo apresenta uma breve revisão atualizada descrevendo a descoberta do sistema SOS, a fisiologia da indução SOS, métodos para sua determinação e o papel de alguns genes induzidos por SOS.
Keywords: Resposta SOS, reparo do DNA, mutações do DNA, reparo propenso a erros, polimerases mutagênicas do DNA
Síntese Histórica
Após ter sido reconhecido que os genes são compostos de DNA (Oswald T. Avery, 1940), numerosos experimentos foram realizados para explorar as propriedades químicas do DNA, principalmente através do tratamento de bactérias e bacteriófagos com uma variedade de agentes e produtos químicos, como luz UV, mitomicina C (MC), etc. Consequentemente, uma lista crescente de mutantes bacterianos mostrando propriedades novas e incomuns foi obtida, e suas propriedades foram posteriormente determinadas.
A hipótese de resposta de SOS foi desenvolvida com base nos seguintes dados: (i) A observação de Jean Weigle em l953 de que a reativação do fago irradiado por raios UV λ aumentou muito quando os fagos irradiados foram plaqueados em células hospedeiras de E. coli previamente irradiadas. Este fenômeno foi posteriormente denominado de reativação W- ou Weigle-; (ii) Indução do profágio λ e lise das bactérias (transformação do desenvolvimento lisogênico em lítico) quando os lisógenos das bactérias E. coli foram irradiados por UV, e (iii) Observação do crescimento filamentoso das células E. coli B em resposta à irradiação UV, sugerindo uma relação entre a parada da divisão celular, os mecanismos de indução do profágio λ e a mutação induzida por UV. . Estes dados levaram Miroslav Radman a concluir que na E. coli existe um sistema de reparação de DNA dependente das proteínas LexA e RecA que é induzido quando o DNA é severamente danificado e sua síntese é presa e sua indução deste sistema está ligada à indução de mutações. Radman chamou-lhe “SOS repair” e “SOS replication” após um sinal de socorro telegráfico (ou óptico) internacional “SOS” no alfabeto Morse (três pontos, três traços, três pontos).
A hipótese SOS de Miroslav Radman foi inicialmente apresentada numa carta inédita enviada a numerosos investigadores em l970, a qual foi posteriormente publicada apenas em 1974 . Evelyn Witkin formulou anteriormente a hipótese de que a formação de filamentos e a indução de profetas nas células E. coli B irradiadas poderiam ter um mecanismo relacionado. A carta original de Radman e o primeiro artigo de Witkin, considerado como a base para a descoberta do fenômeno da resposta SOS, foram recentemente reimpressos em um artigo de Bryn A. Bridges . Outros trabalhos ao longo desta linha confirmaram e desenvolveram esta hipótese. Sistemas que se assemelham em algum aspecto à resposta SOS descrita em E. coli foram mais tarde encontrados para operar também em células eucarióticas, mas as respostas bacterianas e eucarióticas são de fato substancialmente diferentes .
Mecanismo de Indução SOS: Papel da Coprotease RecA* e da Proteína Repressora LexA
Os genes recA e lexA foram os primeiros a serem reconhecidos como estando envolvidos na indução de SOS. As mutações nestes genes tornam as células altamente sensíveis à irradiação UV. As proteínas 27 kDa LexA e 36 kDa RecA eram anteriormente conhecidas como proteínas recombinatórias operando na vida sexual e troca genética de bactérias . Actualmente, sabe-se que a proteína RecA também participa na troca de DNA genético, no recF, recO, recR, recN e na reparação do DNA recombinacional dependente do ruvABC e, juntamente com a proteína LexA, desempenha um papel importante na regulação da resposta SOS. A regulação para baixo e para cima dos genes induzidos por SOS é basicamente uma interação de duas proteínas, LexA repressor e RecA* onde LexA é uma proteína repressora transcripcional, e RecA* é uma coprotease que ajuda a autocatalisação do LexA .
Agentes capazes de induzir o sistema de resposta SOS são, por exemplo radiação UV, MC, metil metanossulfonato (MMS), e muitas outras substâncias químicas que perturbam o ADN, prendem a síntese de ADN e a divisão celular, e levam à acumulação de ADN isolado (ss). O nível de proteína RecA nas células bacterianas (como o da UvrD helicase II) é muito alto. A proteína RecA tem uma forte tendência para formar filamentos de nucleoproteínas no ssDNA, e uma muito mais fraca com DNA quebrado, duplamente encalhado (ds). Isto provavelmente protege o DNA contra a destruição, e é necessário para todos os aspectos da atividade RecA. A montagem de RecA no ssDNA procede na direção 5′-3′ a uma proporção de 1 molécula RecA por 3 bases de DNA, e requer dATP ou ATP, mas sem atividade ATP-ase. A desmontagem, em contraste, requer hidrólise de ATP para ADP e procede muito mais lentamente do que a montagem. RecA montado no ssDNA adquire uma atividade de coprotease, RecA*, que facilita a auto-clevagem da proteína LexA resultando na desrepressão dos genes regulados por SOS. A proteína LexA tem uma fraca atividade de auto-cleavagem, mas sua clivagem e deserepressão dos genes SOS ocorrem apenas na presença da co-protease RecA*.
Cada um dos genes SOS induzidos por damage-induzível (din) ou sos tem perto do seu sítio promotor/operador uma “SOS-box” específica de 20-nucleótidos (também chamada, LexA-box) à qual a proteína repressora LexA está ligada, impedindo a ligação do RNA polimerase e a expressão do gene . A caixa SOS tem uma estrutura palindrómica sugerindo que o repressor LexA se liga como um dímero, como foi posteriormente confirmado . O papel da coprotease RecA* nas células induzidas por SOS é, portanto: 1. ajudar na clivagem da proteína LexA (202 aminoácidos) no local Ala84-Gly85, que causa a deserepressão de SOS-genes ; 2. para clivar o repressor de CI de λ lambda phage, que transforma o fago de uma forma lisogênica para uma forma lítica ; 3. para processar o UmuD → UmuD’, nicking UmuD no site Cys24-Gly25 que é um pré-requisito para a montagem da DNA polimerase V (Pol V) mutagênica induzida por SOS que consiste em UmuD’2C. A etapa limitadora da síntese do Pol V é UmuD→ O processamento da UmuD, que ocorre muito mais lentamente do que a auto-pulverização do LexA. O papel do Pol V na mutagénese é a síntese de translação (TLS) através dos danos no ADN modelo, permitindo a replicação do ADN, frequentemente à custa da fidelidade que leva à mutação. Todas estas proteínas, o repressor CI do λ fago e repressor LexA, UmuD, PolB/DinA (Pol II) e proteínas DinB (Pol IV) são homólogos dentro de seus domínios carboxi-terminais, e todas são codificadas por genes din (sos) regulados sob resposta SOS.
Indução da resposta SOS prossegue até 45-60 min após o tratamento de bactérias com agentes indutores de SOS e, em seguida, cessa abruptamente. Dentro deste tempo a maioria das lesões já foi reparada. O tempo de desrepressão dos genes individuais do din depende da força da ligação do repressor LexA com a caixa SOS e da facilidade com o repressor LexA é separada de uma caixa SOS específica.
3.1. Por din::lacZ formation e β-galactosidase assay
A resposta SOS foi estudada anteriormente testando o aumento da expressão dos genes din a partir dos genes naturais, ou usando uma construção de um gene repórter, por exemplo, fundindo um promotor din putativo com o gene promotor-less lacZ codificando β-galactosidase. Graham Walker e colegas de trabalho foram os primeiros a empregar para esta tarefa um fago defeituoso, Mu1d(Ap,lacZ) construído por Casadaban e Cohen , que facilmente insere aleatoriamente no cromossoma de E. coli K12 e cria uma mutação. Este fago carrega um gene lacZ sem promotor, de modo que β-galactosidase não é expresso. Entretanto, quando o fago Mu é por acaso integrado sob o promotor de um gene din formando um din funcional::Mu-1d(Ap,lacZ) operon, a β-galactosidase é sintetizada em resposta a danos no DNA.
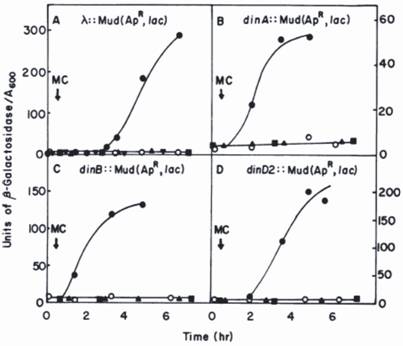
Desde a hidrólise do substrato β-galactosidase (o-nitrofenil-β-galactoside) forma colónias amarelas, as que contêm din::Mu-lacZ fusion são facilmente seleccionadas em placas de ágar; o nível preciso de β-galactosidase expresso em resposta a danos no ADN pode então ser medido com precisão em meio líquido (ver Fig.1 para detalhes). Desta forma mais de dez novos din-genes foram detectados e a maioria deles posteriormente identificados.
Cinética de indução de β-galactosidase em din::lacZ cepas de fusão por mitomicina C (MC) . As fusões din::lac foram geradas pela inserção do bacteriófago Mu d1(Ap, lac) no cromossomo E. coli. A derivada λ::Mu d1(Ap lac) foi gerada por uma inserção de Mu d1(Ap lac) no fago λ no cromossomo E. coli. Símbolos: o, cepa de fusão não tratada; ●, cepas de fusão mais MC; derivados lexA(Ind-) da cepa de fusão mais MC; ■, derivados recA (Def) da cepa de fusão mais MC; ▼, um derivado contendo pKM280 da λ:: estirpe Mu d1(Ap lac) mais MC. Reimpresso com a permissão do autor. Dois dos genes, dinA e dinB foram posteriormente identificados como polB (Pol II) e Pol IV, respectivamente .
 (Clique na imagem para ampliar.)
(Clique na imagem para ampliar.)
Recentemente, um novo método foi elaborado para medir a expressão do gene SOS e a atividade promotora dos genes SOS (por exemplo recA, lexA, umuDC) usando um plasmídeo contendo um promotor SOS a ser investigado fundido ao gene repórter proteína fluorescente verde codificadora gfp (GFP) . Isto permite medir a actividade promotora dos genes SOS numa única célula bacteriana, assim como a localização e duração da indução SOS. Parece que a indução dos genes SOS não procede como um único evento, mas segue em várias etapas repetíveis cuja modulação depende da dose indutora de SOS, do nível de dano no DNA e do acúmulo de proteína UmuD’. Este método abre uma nova forma de medir a dinâmica da resposta SOS.
3.2. Procurando por caixas SOS: Determinação do índice de heterologia (HI)
Progresso na sequenciação do ADN e conhecimento dos elementos característicos das sequências de genes SOS permitiu pesquisas computacionais directas de genes induzíveis por SOS. Quando 33% do DNA E. coli cromossômico havia sido sequenciado, Lewis et al. localizaram por análise de seqüências e experimentos quantitativos de ligação de DNA seis novos genes potencialmente regulados por LexA, e os chamaram de sosA-F . Para dois deles, sosC e sosD, os autores confirmaram experimentalmente que eles ligavam fortemente o repressor LexA purificado.
Subsequentemente, ao comparar as sequências de caixas SOS de 19 din genes conhecidos na altura (incluindo sosA-sosF), estabeleceram que a sequência consensual de caixas SOS é um palíndromo perfeito, TACTG(TA)5CAGTA ; e com base na teoria de Berg e von Hippel calcularam matematicamente para cada uma das caixas SOS um índice de heterologia (HI). Este índice indica o desvio de uma caixa SOS do consenso e, quando o seu valor é baixo, o gene é fortemente suprimido, e quando o seu valor é alto é mais facilmente descomprimido. Em um HI maior que 15, o repressor LexA não se liga à caixa SOS. Portanto, o valor HI é uma medida da força relativa do repressor LexA ligado a uma determinada caixa SOS, e é responsável pela variação do potencial de desrepressão.
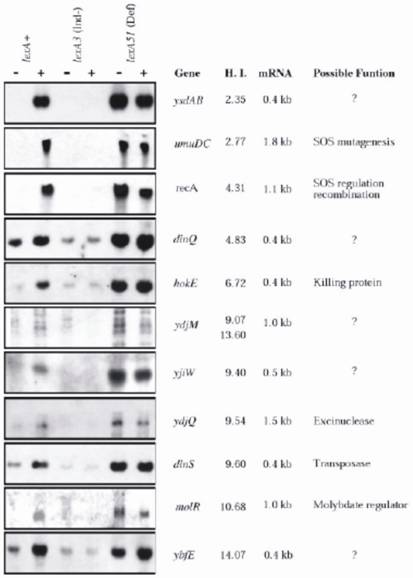
Quando a seqüência de DNA de todo o cromossomo E. coli K12 foi determinada, Fernandez de Henestrosa et al localizou, procurando por potenciais caixas SOS associadas a quadros de leitura abertos, 69 potenciais caixas SOS com um valor HI ≤ 15, incluindo todas as previamente conhecidas e sete romances. Os novos genes foram posteriormente analisados quanto à sua capacidade de serem expressos no tratamento de MC, quanto ao comprimento do mRNA expresso e quanto aos valores de HI (Fig. 2). As análises foram realizadas em três linhagens isogênicas de E. coli diferindo no alelo lexA: lexA+(tipo selvagem) SOS-induzível, SOS não-induzível lexA3(Ind-), e o alelo lexA51(Def) constitutivamente expresso. Potenciais funções dos genes novo e antigo foram ainda mais caracterizadas; e discutidas. Os resultados confirmaram que cada um dos novos genes contendo caixa SOS era de fato um gene dependente da LexA, e sua expressão foi induzida pela MC apenas na cepa lexA+; caso contrário, ou não foram expressos (lexA3), ou totalmente expressos (lexA51), independentemente de as bactérias terem sido ou não tratadas com MC (veja a Fig. 2 para detalhes).
Da sequência nucleotídica do gene ydjQ (nomes alternativos b1741 sosD) deduziu-se que codifica a proteína 295 aminoácidos que partilha uma homologia significativa com a metade terminal da proteína N -terminal da UvrC . A UvrABC-excinuclease (que consiste em UvrA, UvrB e proteínas UvrC) era conhecida por estar envolvida na reparação de excisão de nucleotídeos (NER) que remove adutos volumosos ou lesões que afetam a estrutura (por exemplo, dímeros de pirimidina, fotoprodutos de pirimidina (6-4) e locais abásicos) do DNA modificado. Sabia-se também que a parte N da UvrC incide o ADN ssDNA inicialmente no lado 3′ da lesão, e depois a parte C da UvrC incide o ADN no lado 5′ da lesão. Recentemente, Moolenaar et al, renomearam a proteína YdjQ como Cho (depois do homólogo da UvrC) e examinaram sua atividade enzimática. Eles confirmaram que a proteína Cho incide complexos de préincisão UvrB-DNA apenas no lado 3′- da lesão; entretanto, algumas lesões no DNA que foram muito mal incisadas pela proteína UvrC foram muito eficientemente incisadas pela proteína Cho. Assim, a proteína Cho aumenta muito a gama de substratos na reparação do ADN pelo sistema NER. Os genes uvrA, uvrB, e ydjQ (mas não uvrC) são genes induzidos por SOS.
Análise norte dos genes E. coli que parecem ser regulados pelo LexA. O RNA foi extraído de três linhagens isogênicas que diferiram no gene lexA: RW118 (lexA+), RW434 (lexA), e RW542 (lexA51). O RNA foi obtido de células não danificadas (-) e de células que tinham sido expostas à mitomicina C (5 µg ml-1) (+) durante 30 min antes da extração. Os genes recA e umuDC previamente identificados foram usados como controles positivos. Os genes são representados de acordo com seu índice de heterologia ascendente (HI). Os tamanhos da transcrição do mRNA e as possíveis funções dos genes também são indicados Veja a ref. para mais detalhes. (Por cortesia da Blackwell Science).
 (Clique na imagem para ampliar.)
(Clique na imagem para ampliar.)
3.3. Localização de genes de din por técnicas de microarray
A técnica de microarray permite um grande número de genes a serem monitorados em um experimento. Courcelle et al., utilizaram microarrays contendo fragmentos cromossómicos de ADN E coli amplificados com quadros de leitura abertos de 4101 genes (95,5% do total) para medir a expressão de todos os genes em estirpes SOS-induzíveis (lexA+) e não-induzíveis (lexA1(Ind-)). Na cepa lexA+ irradiada por UV, os autores identificaram 17 novos genes induzidos por SOS dependentes de LexA, além dos 26 conhecidos anteriormente; portanto, o número total de genes induzíveis por SOS na E. coli é provavelmente 43. Na mesma publicação, os autores estabeleceram que a codificação do gene ssb para uma proteína de ligação SsDNA- não é induzível por SOS, como já foi pensado anteriormente. Eles também observaram um número de genes cuja expressão aumentou (geralmente não acima de duas vezes) em células irradiadas por UV, mas que não foram reguladas pela proteína LexA. Observaram que as transcrições proteicas de muitos genes não regulados por LexA foram reduzidas após a radiação UV, e concluíram que essas transcrições provavelmente foram danificadas ou degradadas pela radiação UV. Eles também identificaram trinta genes com estruturas tipo caixa SOS em potencial, mas que não foram regulados pelo LexA.
Mecanismo e Especificidade da Ligação do Repressor LexA às Caixas SOS
Acredita-se que as seqüências de todas as caixas SOS em potencial no cromossomo E. coli foram identificadas. Alguns exemplos de caixas SOS que ligam (A) ou não ligam (B e C) repressor LexA, juntamente com os valores HI e o número de desajustes (NM) são mostrados na Tabela 1. NM denota o número de posições nas caixas SOS que se desviam de um palíndromo perfeito. Tanto o número como o padrão de desencontros podem ser fundamentais para a especificidade da ligação da proteína LexA a cada caixa SOS individual. Parece que esta hipótese é uma boa explicação para a especificidade e força diferente da ligação da proteína LexA a sequências de caixas SOS. Mas isto deve ser confirmado.
Pode-se ver que geralmente, quando as caixas SOS têm baixos valores de HI, entre 2,7 e 12, elas podem ligar o repressor LexA (Tabela 1A); e quando a HI está acima de 16,4 (Tabela 1B) elas aparentemente são incapazes de ligar o repressor LexA. Entretanto, em alguns casos (mostrado na parte C) como o yigN (nome alternativo sosB), e dinJ (sosA), as caixas SOS não conseguem ligar o repressor LexA apesar de seus valores moderados de HI (9.27 e 7.06, respectivamente) .
Potenciais caixas SOS de genes que ligam ou não ligam o repressor LexA.
| Gene | Sequência de caixas SOS | HI* | NM** |
|---|---|---|---|
| Consenso | TACTGTATATATATACAGTA | 0 | |
| A. Genes cujas caixas SOS ligam o LexA e são reguladas pelo representante LexA | |||
| recA | TACTGTATGAGCATACAGTA | 4.31 | 1 |
| umuDC | TACTGTATATAAAAACAGTA | 2.77 | 2 |
| uvrB | AACTGTTTTTTTATCCAGTA | 6.11 | 5 |
| polB | GACTGTATAAAACCACAGCC | 12.09 | 5 |
| lexA1 | TGCTGTATATACTCACAGCA | 6.34 | 4 |
| lexA2 | AACTGTATATACACCCAGGGG | 8.32 | 6 |
| B. Genes cujas caixas SOS potenciais não ligam LexA mas não são reguladas por LexA | |||
| intE | GCTGCTGAAAAATACAGAA | 16.04 | 7 |
| ymfI | TTCTGTACCAGAAAACAGTTT | 15.48 | 8 |
| ymfM | AGCTGCAGGAGCATGCAGCA | 19.32 | 3 |
| lit | TGATGACAGTGTCCAGTG | 20.32 | 8 |
| C. Genes cujas caixas SOS não ligam LexA em série de baixo valor HI | |||
| yigN | AACTGGACGTTTGTACAGCA | 9,27 | 5 |
| dinJ | AGCTGAATAAATACAGCA | 7.06 | 3 |
Potenciais caixas SOS (sequência no fio de codificação) que ligam (A), ou não ligam (B e C) repressor LexA.
HI* denota o índice de heterologia; NM*** denota o número de desencontros em caixas SOS que se desviam de um palíndromo perfeito. A falta de ligação do repressor LexA apesar de um valor HI relativamente baixo (secção C) atesta que não existe uma correlação directa entre eles . De qualquer forma, indica que o valor HI não pode ser o único indicador da capacidade de uma caixa SOS de ligar o LexA. O número de desajustes nas caixas SOS palíndromas em cada uma das secções é semelhante, e portanto não determina a capacidade de ligação LexA com as caixas SOS. Os dados nas partes A e C são da ref. Os dados na parte B são da ref. .
Características de Alguns Genes Induzidos por SOS
Os genes de resposta SOS são encontrados espalhados pelo cromossoma E. coli como genes únicos situados em óperos únicos. Seis deles, umuDC (a fonte do Pol V), ruvAB (migração de ramo catalisador nas estruturas de Holliday), e ysdAB (de função desconhecida) são codificados por pares de genes formando um ópero. Geralmente, apenas uma caixa SOS está presente em um ópero. As exceções são os genes lexA e ydjM (b1728) que contêm duas caixas SOS cada uma (separadas por uma e duas bases, respectivamente) e recN contendo três caixas SOS. As seqüências das caixas SOS em um gene são diferentes . No caso do ydjM, dois repressores diméricos LexA ligam-se cooperativamente a cada caixa SOS, e como estimado, ambos são funcionais . As seqüências das caixas SOS em um gene diferem em 2 a 4 bases. Como e porque as caixas SOS extras nos genes surgem, e como elas influenciam o potencial de expressão gênica são questões que ainda não foram respondidas.
O tempo necessário para aerepressão dos genes induzidos por SOS
A escala de tempo para a deserepressão gênica e síntese das proteínas induzidas por SOS varia para os genes individuais. Os genes mais rapidamente desreprimidos (<1 min depois da indução de SOS) incluem: lexA codificando a proteína repressora LexA (rapidamente degradada nas células induzidas por SOS), uvrAB, cho e uvrD envolvidas no reparo do NER, ruvAB participando no reparo do DNA recombinacional, polB e dinB codificando Pol II e Pol IV, respectivamente , e dinI, cujo produto inibe o processamento de UmuD a UmuD’ . A proteína UmuD’ é necessária para a síntese do Pol V mutagênico (UmuD’2C) . Portanto, a proteína DinI retarda a síntese de Pol V. A expressão dos genes recA e recN que codificam as proteínas de recombinação e reparação recombinacional, ocorre 5 minutos após a indução de SOS, enquanto a de sulA (nome antigo, sfiA) e umuDC ocorre no último estágio da indução de SOS . A proteína SulA é um inibidor da divisão celular causando o crescimento filamentoso das células e prolongando o tempo durante o qual o DNA celular pode ser reparado.
Copy Numbers of din Genes Encoding Proteins
RecA e UvrD pertencem às proteínas sintetizadas mais abundantemente. Os seus números a nível constitutivo são, respectivamente, cerca de 10.000 e 8.000 cópias por célula e aumentam 10 vezes após a indução de SOS . A proteína RecA liga-se ao ssDNA e provavelmente protege-o contra a destruição descontrolada. O UvrD, DNA helicase II, participa no reparo da incompatibilidade entre o SsDNA e o SsDNA dependente de barragem (MMR) e participa no reparo do UvrABC e do SsDNA dependente de Choque (NER), deslocando o SsDNA que contém ssDNA da cadeia de DNA reparado. Os números de moléculas proteicas sintetizadas em células não induzidas vs. células induzidas por SOS são os seguintes: 20:250 para UvrA; 250:1000 para UvrB; 40:300 para DNA Pol II, e 250:2500 para DNA Pol IV (11, 34). A proteína UmuD é expressa, a 180 moléculas por célula repressora não induzida, e a 2400 moléculas por célula repressora não funcional LexA; há 200 moléculas UmuC por célula induzida por SOS e nenhum Pol V (< 15 moléculas) em célula não induzida .
Polimerases de DNA Mutagênicas induzidas por SOS
Na E. coli, além do DNA sintetizado constitutivamente replicando Pol III há três polimerases de DNA potencialmente mutagênicas cuja síntese é aumentada (Pol II e Pol IV) ou ocorre apenas em células induzidas por SOS (Pol V). Entre estas, Pol II é a única DNA polimerase que possui uma atividade de leitura de 3′-5′ exonuclease proofreading e é a menos propensa a erros; seu papel também inclui a recuperação de DNA degradado em garfos de replicação . Tanto o pol II como o pol IV aparecem nas fases iniciais da indução de SOS, e o pol V na sua fase final. Pol V é a enzima mais propensa a erro e a mais importante para a mutagenicidade das células induzidas por SOS.
Na E. coli defeituosa em umuD(C) quase nenhuma mutação é induzida após a irradiação UV e as mutações induzidas pelo tratamento MMS são grandemente reduzidas . As principais lesões mutagênicas formadas no DNA após a irradiação UV são os dímeros TT-cis-syn cyclobutane e os fotoprodutos de TC ou TT (6-4) , enquanto que após o tratamento com MMS, 3-metiladenina (3meA), locais apurínicos e 1meA e 3meC (em células alkB-mutantes) são encontrados predominantes. Ambos UV e MMS, como muitos outros mutagênios, são indutores de SOS e, portanto, embora as lesões prejudiciais sejam diferentes, o sinal indutor de SOS deve ser comum; é geralmente aceito que os sinais indutores de SOS são filamentos RecA/ssDNA formados no acúmulo de ssDNA nas células quando a síntese de DNA é presa. Algumas das lesões pré-mutagênicas requerem polimerases de DNA mutagênicas para levar a mutações, enquanto outras não. Entretanto, qual a polimerase de DNA mutagênica induzida por SOS depende do tipo de lesão .
Conclusão
A hipótese de resposta de SOS foi surpreendente, frutífera e inspiradora. Reunimos muitas informações sobre o metabolismo do DNA, a expressão dos genes induzidos por SOS, e suas funções. No entanto, novas idéias ainda estão por vir.
Agradecimentos
Estou muito grato ao Professor Graham C. Walker, Roger Woodgate e Blackwell Science Publisher pela permissão de reimpressão.
Conflito de Interesses
O autor declarou que não há conflito de interesses.
1. Weigle JJ. Indução de mutação em um vírus bacteriano. Proc Natl Acad Scien U S A. 1953;39(7):628-636
2. Radman M. Phenomenology of an inducible mutagenic DNA repair pathway in Escherichia coli: SOS repair hypothesis. In: (ed.) Sherman S, Miller M, Lawrence C, Tabor WH. Aspectos Moleculares e Ambientais da mutagénese. Springfield IL: Charles C Thomas, editor. 1974:128-142
3. Borek E, Ryan A. The transfer of irradiation-elicited induction in a lysogenic organism. Proc Natl Acad Scientifie U S A. 1958;44(5):374-347
4. Hertman I, Luria SE. Transduction studies on the role of a rec+ gene in the ultraviolet induction of prophage lambda. J Mol Biol. 1967;23(2):117-133
5. Defais M, Fauquet P, Radman M, Errera M. Reativação ultravioleta e mutagênese ultravioleta da lambda em diferentes sistemas genéticos. Virologia. 1971;43(2):495-503
6. Craig R. Função do trifosfato nucleosídeo e polinucleotídeo em Escherichia coli recA clivagem protéica de fago repressor lambda. J Biol Chem. 1981;256(15):8039-8044
7. Witkin EM. Mutação induzida por ultravioleta e reparação do ADN. Annu Rev Microbiol. 1969;23:487-514
8. Bridges BA. Reparação de ADN susceptível de erro e síntese de ADN de translação II: A hipótese de SOS induzível. Reparação do ADN (Amst). 2005;4(6):725-739
9. Eller MS, Asarch A, Gilchrest BA. Fotoprotecção na pele humana – uma resposta SOS multifacetada. Phtochem & Photobiol. 2008;84:339-349
10. Clark AJ, Margulies AD. Isolamento e caracterização de mutantes com deficiência de recombinação de Escherichia coli K12. Proc Natl Acad Scien U S A. 1965;53:451-459
11. Kuzminov A. Reparação recombinacional de danos de DNA em Escherichia coli e bacteriófagos lambda. Microbiol Mol Biol Rev. 1999;63(4):751-813
12. Horii T, Ogawa T, Nakatani T, Hase T, Matsubara H, Ogawa H. Regulação das funções SOS: purificação da proteína E. coli LexA e determinação do seu local específico clivado pela proteína RecA. Célula. 1981;27(3 Pt 2):515-522
13. Pequeno JW, Monte DW. O sistema regulador SOS da Escherichia coli. Célula. 1982;29(1):11-22
14. Walker GC. Mutagênese e respostas induzíveis aos danos causados pelo ácido desoxirribonucleico na Escherichia coli. Microbiol Rev. 1984;48(1):60-93
15. Arenson TA, Tsodikov OV, Cox MM. Análise quantitativa da cinética da desmontagem de filamentos RecA de ssDNA dependente do fim. J Mol Biol. 1999;288(3):391-401
16. Schlacher K, Pham P, Cox MM, Goodman MF. Roles of DNA polimerase V and RecA protein in SOS damage-induced mutation. Chem Rev. 2006;106(2):406-419
17. Lewis LK, Harlow GR, Gregg-Jolly LA, Mount DW. Identificação de locais de ligação de alta afinidade para LexA que definem novos genes induzíveis por danos de DNA na Escherichia coli. J Mol Biol. 1994;241(4):507-523
18. Thliveris AT, Little JW, Mount DW. A repressão do gene E coli recA requer pelo menos dois monômeros de proteína LexA. Biochimie. 1991;73(4):449-456
19. Burckhardt SE, Woodgate R, Scheuermann RH, Echols H. UmuD mutagenesis protein of Escherichia coli: overproduction, purification, and cleavage by RecA. Proc Natl Acad Scien U S A. 1988;85(6):1811-1815
20. Shinagawa H, Iwasaki H, Kato T, Nakata A. RecA clivagem dependente de proteína UmuD e mutagênese de SOS. Proc Natl Acad Sci EUA. 1988;85(6):1806-1810
21. Tang M, Shen X, Frank EG, O’Donnell M, Woodgate R, Goodman MF. UmuD'(2)C é uma DNA polimerase propensa a erros, Escherichia coli pol V. Proc Natl Acad Scien U S A. 1999;96(16):8919-8924
22. Kenyon CJ, Walker GC. Os agentes nocivos ao DNA estimulam a expressão gênica em loci específicos em Escherichia coli. Proc Natl Acad Sci U S A. 1980;77(5):2819-2823
23. Casadaban MJ, Cohen SN. Genes de lactose fundidos a promotores exógenos em uma única etapa usando uma bacteriófago Mu-lac: sonda in vivo para seqüências de controle transcripcionais. Proc Natl Acad Sci U S A. 1979;76(9):4530-4533
24. Bonner CA, Hays S, McEntee K, Goodman MF. O DNA Polimerase II é codificado pelo gene dinA do DNA danificável da Escherichia coli. Proc Natl Acad Sci USA. 1990;87:7663-7667
25. Wagner J, Gruz P, Kim SR, Yamada M, Matsui K, Fuchs RPP, Nohmi T. O gene dinB codifica uma nova E. coli DNA polimerase, DNA pol IV, envolvida em mutagênese. Célula Mol. 1999;4(2):281-286
26. Friedman N, Vardi S, Ronen S, Alin M, Stavans J. Precise temporal modulation in the response of the SOS DNA repair network in individual bacteria. PLoS Biol. 2005;3(7):e238
27. Krishna S, Maslov S, Sneppen K. Mutagénese induzida por UV na resposta de Escherichia coli SOS: um modelo quantitativo. PLoS Comput Biol. 2007;3(3):e41
28. Fernandez de Henestrosa AR, Ogi T, Aoyagi S, Chafin D, Hayes JJ, Ohmori H, Woodgate R. Identificação de genes adicionais pertencentes ao regulador LexA na Escherichia coli. Mol Microbiol. 2000;35(6):1560-1572
29. Selby CP, Sancar A. Mecanismos de acoplamento transcrição-reparação e declínio da frequência de mutação. Microbiol Rev. 1994;58(3):317-29
30. Moolenaar GF, van Rossum-Fikkert S, van Kesteren M, Goosen N. Cho, uma segunda endonuclease envolvida na reparação da excisão de Escherichia coli nucleotide. Proc Natl Acad Scien U S A. 2002;99(3):1467-1472
31. Courcelle J, Khodursky A, Peter B, Brown PO, Hanawalt PC. Perfis comparativos de expressão gênica após exposição UV em Escherichia coli de tipo selvagem e SOS-deficiente. Genética. 2001;158(1):41-64
32. Yasuda T, Morimatsu K, Horii T, Nagata T, Ohmori H. Inhibition of Escherichia coli RecA coprotease by Din I. EMBO J. 1998;17(11):3207-3216
33. Cooper DL, Lahue R.S, Modrich P. Mismatch repair in replication fidelity, genetic recombination, and cancer biology. Annu Rev Biochem. 1996;65:101-133
34. Kim S.-R, Matsui K, Yamada M, Gruz P, Nohmi T. Papéis dos genes cromossômicos que codificam o DNA pol IV na mutagênese direcionada e não direcionada em Escherichia coli. Célula Mol. 1999;4(2):281-286
35. Woodgate R, Ennis DG. Níveis de proteínas Umu codificadas cromossomicamente e requisitos para clivagem in vivo UmuD. Mol Genet. 1991;229(1):10-16
36. Rangarajan S, Woodgate R, Goodman MF. Um fenótipo para DNA polimerase II enigmático em reinício de replicação em Escherichia coli irradiada por raios UV. Proc Natl Acad Sci USA. 1999;96(16):9224-9229
37. Kato T, Shinoura Y. Isolamento e caracterização de mutantes de Escherichia coli deficiente na indução de mutações por luz ultravioleta. Mol Genet. 1977;156(2):121-131
38. Sledziewska-Gojska E, Janion C. Caminhos alternativos da mutagénese induzida pelo metanossulfonato de metilo na Escherichia coli. Mol Genet. 1989;216(1):126-31
39. Nieminuszczy J, Sikora A, Wrzesinski M, Janion C, Grzesiuk E. AlkB dioxygenase na prevenção da mutagénese induzida por MMS na Escherichia coli: efeito das proteínas Pol V e AlkA. Reparação do ADN (Amst). 2006;5(2):181-188
40. Napolitano R, Janel-Bintz R, Wagner J, Fuchs RP. Todas as três polimerases de DNA induzíveis por SOS (Pol II, Pol IV e Pol V) estão envolvidas na mutagénese induzida. EMBO J. 2000;19(22):6259-6265
41. Fuchs RP, Fujii S, Wagner J. Propriedades e funções da Escherichia coli: Pol IV e Pol V. Adv Protein Chem. 2004;69:229-264
42. Foster PL. Mutagênese induzida por estresse em bactérias. Crit Rev Biochem Molec Biol. 2007;42:373-397
43. Iwasaki H, Nakata A, Walker GC, Shinagawa H. O gene Escherichia coli polB, que codifica a DNA polimerase II, é regulado pelo sistema SOS. J Bacteriol. 1990;172(11):6268-6273