Int J Biol Sci 2008; 4(6):338-344. doi:10.7150/ijbs.4.338
Review
Celina Janion ![]()
Institute of Biochemistry and Biophysics, Polish Academy of Sciences, Pawinskiego 5a, 02-106 Warszawa, Polen
Janion C. Inducible SOS Response System of DNA Repair and Mutagenesis in Escherichia coli. Int J Biol Sci 2008; 4(6):338-344. doi:10.7150/ijbs.4.338. Tilgængelig fra https://www.ijbs.com/v04p0338.htm
Chromosomalt DNA er udsat for løbende beskadigelse og reparation. Cellerne indeholder en række proteiner og specifikke DNA-reparationssystemer, som hjælper med at opretholde dets korrekte struktur. SOS-reaktionen var det første DNA-reparationssystem, der blev beskrevet i Escherichia coli, og som blev induceret ved behandling af bakterier med DNA-skadelige stoffer, der standser DNA-replikation og celledeling. Induktion af SOS-reaktionen involverer mere end fyrre uafhængige SOS-gener, hvoraf de fleste koder for proteiner, der er involveret i beskyttelse, reparation, replikation, mutagenese og metabolisme af DNA. Under normale vækstbetingelser udtrykkes SOS-generne på et basalt niveau, som stiger markant ved induktion af SOS-responset. SOS-responset er fundet i mange bakteriearter (f.eks. Salmonella typhimurium, Caulobacter crescentus, Mycobacterium tuberculosis), men ikke i eukaryote celler. Arter fra alle riger indeholder imidlertid nogle SOS-lignende proteiner, der deltager i DNA-reparation, og som udviser aminosyrehomologi og enzymatiske aktiviteter, der er beslægtet med dem, der findes i E. coli, men som ikke er organiseret i et SOS-system. Denne artikel præsenterer en kort opdateret gennemgang, der beskriver opdagelsen af SOS-systemet, fysiologien ved SOS-induktion, metoder til bestemmelse heraf og den rolle, som nogle SOS-inducerede gener spiller.
Nøgleord: SOS-reaktion, DNA-reparation, DNA-mutationer, fejlpræget reparation, mutagene DNA-polymeraser
Historisk oversigt
Når det blev erkendt, at gener består af DNA (Oswald T. Avery, 1940), blev der udført talrige eksperimenter for at udforske DNA’s kemiske egenskaber, hovedsagelig ved at behandle bakterier og bakteriofager med en række midler og kemikalier, som f.eks. UV-lys, mitomycin C (MC) osv. Som følge heraf blev der opnået en voksende liste af bakteriemutanter, der viste nye og usædvanlige egenskaber, og deres egenskaber blev efterfølgende bestemt.
Hypotesen om SOS-respons blev udviklet på grundlag af følgende data: (i) Jean Weigles iagttagelse i l953, at reaktiveringen af UV-bestrålet fage λ øgedes kraftigt, når de bestrålede fager blev udplottet på tidligere bestrålede E. coli-værtsceller. Dette fænomen blev senere betegnet W- eller Weigle-reaktivering ; (ii) Induktion af profage λ og lysering af bakterier (transformation fra lysogen til lytisk udvikling), når E. coli-bakterielysogener blev UV-bestrålet , og (iii) Observation af filamentøs vækst af E. coli B-celler som reaktion på UV-bestråling, hvilket tyder på en sammenhæng mellem stop af celledelingen, mekanismerne for λ-profageinduktion og UV-induceret mutation. . Disse data fik Miroslav Radman til at konkludere, at der i E. coli findes et DNA-reparationssystem, der er afhængigt af LexA- og RecA-proteinerne, som induceres, når DNA er alvorligt beskadiget og dets syntese standses, og at induktionen af dette system er forbundet med induktion af mutationer. Radman kaldte det “SOS-reparation” og “SOS-replikation” efter et internationalt telegrafisk (eller optisk) nødsignal “SOS” i Morse-alfabetet (tre prikker, tre streger, tre prikker).
Miroslav Radmans SOS-hypotese blev oprindeligt fremsat i et upubliceret brev sendt til en lang række forskere i l970, som efterfølgende først blev offentliggjort i1974 . Evelyn Witkin opstillede tidligere den hypotese, at dannelsen af filamenter og profageinduktion i bestrålede E. coli B-celler kunne have en beslægtet mekanisme. Radmans oprindelige brev og Witkins tidlige artikel, der betragtes som grundlaget for opdagelsen af SOS-responsfænomenet, er for nylig blevet genoptrykt i en artikel af Bryn A. Bridges . Yderligere arbejde i denne retning bekræftede og udviklede denne hypotese. Systemer, der i nogen henseende ligner det SOS-respons, der er beskrevet i E. coli, blev senere fundet at fungere også i eukaryote celler, men de bakterielle og eukaryote responser er faktisk væsentligt forskellige.
Mekanismen for SOS-induktion: Rolle af RecA* coprotease og LexA-repressorprotein
Generne recA og lexA var de første, der blev anerkendt som værende involveret i SOS-induktion. Mutationer i disse gener gør cellerne meget følsomme over for UV-stråling. De 27 kDa LexA- og 36 kDa RecA-proteiner var tidligere kendt som rekombinationsproteiner, der opererer i bakteriers seksuelle liv og genetiske udveksling . I dag ved man, at RecA-proteinet også deltager i genetisk DNA-udveksling, i recF-, recO-, recR-, recN- og ruvABC-afhængig rekombinations-DNA-reparation , og sammen med LexA-proteinet spiller det en vigtig rolle i reguleringen af SOS-responset. Ned- og opregulering af de SOS-inducerede gener er grundlæggende et samspil mellem to proteiner, LexA-repressor og RecA*, hvor LexA er et transkriptionelt repressorprotein, og RecA* er en coprotease, der hjælper den autokatalytiske selvspaltning af LexA .
Agenser, der er i stand til at inducere SOS-respons-systemet, er f.eks, UV-stråling, MC, methylmethansulfonat (MMS) og mange andre kemikalier, der forstyrrer DNA, standser DNA-syntesen og celledelingen og fører til ophobning af enkeltstrenget (ss) DNA. Niveauet af RecA-protein i bakterieceller (ligesom niveauet af UvrD helicase II) er meget højt. RecA-proteinet har en stærk tendens til at danne nukleoproteinfilamenter på ssDNA og en meget svagere tendens med brudt, dobbeltstrenget (ds) DNA . Dette beskytter sandsynligvis DNA mod ødelæggelse, og er nødvendigt for alle aspekter af RecA-aktiviteten. Samlingen af RecA på ssDNA foregår i 5′-3′-retningen i et forhold på 1 molekyle RecA pr. 3 DNA-baser og kræver dATP eller ATP, men ingen ATP-aseaktivitet. Afmonteringen kræver derimod hydrolyse af ATP til ADP og forløber meget langsommere end samlingen. RecA, der er samlet på ssDNA, erhverver en coproteaseaktivitet, RecA*, som letter selvspaltningen af LexA-proteinet, hvilket resulterer i derepression af SOS-regulerede gener. LexA-proteinet har en svag selvspaltningsaktivitet, men dets spaltning og derepression af SOS-generne sker kun i tilstedeværelse af RecA*-koprotease.
Hver af de SOS-inducerede skade-inducerbare (din) eller sos-gener har i nærheden af deres promotor/operatorsted en specifik 20-nucleotid-lang “SOS-box” (også kaldet LexA-box), som LexA-repressorproteinet er bundet til, hvilket forhindrer RNA-polymerase-binding og genekspression . SOS-boksen har en palindromisk struktur, hvilket tyder på, at LexA-repressoren binder som en dimer, hvilket senere blev bekræftet . RecA*-koproteasen i SOS-inducerede celler har derfor følgende rolle: 1. at hjælpe med at kløve LexA-proteinet (202 aminosyrer) på Ala84-Gly85-stedet, hvilket medfører derepression af SOS-generne ; 2. at hjælpe med at kløve LexA-proteinet (202 aminosyrer) på Ala84-Gly85-stedet, hvilket medfører derepression af SOS-generne ; 3. at hjælpe med at kløve LexA-proteinet (202 aminosyrer) på Ala84-Gly85-stedet. at kløve CI-repressoren i λ lambda-fagen, hvilket omdanner fagen fra en lysogen til en lytisk form ; 3. at behandle UmuD → UmuD’ ved at knække UmuD på Cys24-Gly25-stedet, hvilket er en forudsætning for samling af den SOS-inducerede mutagene DNA-polymerase V (Pol V), der består af UmuD’2C. Det hastighedsbegrænsende trin i Pol V-syntesen er UmuD→ UmuD’-processering, som sker meget langsommere end LexA’s selvspaltning. Pol V’s rolle i mutagenese er translesionssyntese (TLS) over skaden i skabelon-DNA, hvilket muliggør DNA-replikation, ofte på bekostning af troværdighed, hvilket fører til mutation . Alle disse proteiner, CI-repressoren for λ-fage og LexA-repressoren, UmuD, PolB/DinA (Pol II) og DinB (Pol IV) proteinerne er homologe inden for deres carboxy-terminale domæner, og alle er kodet af din (sos) gener, der reguleres under SOS-respons.
Induktion af SOS-respons fortsætter indtil 45-60 min efter behandling af bakterier med SOS-inducerende midler og ophører derefter pludseligt. Inden for dette tidsrum er de fleste læsioner blevet repareret. Timingen af derepressionen af de enkelte din-gener afhænger af styrken af LexA-repressorens binding med SOS-boksen og af, hvor let LexA-repressoren løsnes fra en bestemt SOS-boks.
3.1. Ved din::lacZ dannelse og β-galactosidase assay
SOS-reaktionen er tidligere blevet undersøgt ved at teste stigningen i din-genernes ekspression enten fra de naturlige gener eller ved at bruge en reportergenkonstruktion, f.eks. fusion af en formodet din-promotor med et promotorløst lacZ-gen, der koder for β-galactosidase. Graham Walker og medarbejdere var de første til at anvende en defekt fage, Mu1d(Ap,lacZ), konstrueret af Casadaban og Cohen , som let kan indsættes tilfældigt i E. coli K12’s kromosom og skabe en mutation, til denne opgave. Denne fage bærer et lacZ-gen uden promotor, således at β-galactosidase ikke udtrykkes. Når Mu-fagen imidlertid ved et tilfælde integreres under promotoren for et din-gen, der danner et funktionelt din::Mu-1d(Ap,lacZ)-operon, syntetiseres β-galactosidase som reaktion på DNA-skader.
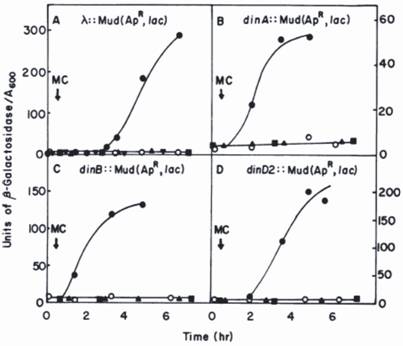
Da hydrolyse af β-galactosidase-substratet (o-nitrophenyl-β-galactosid) danner gule kolonier, kan de kolonier, der bærer en din::Mu-lacZ-fusion, let udvælges på agarplader; det præcise niveau af β-galactosidase, der udtrykkes som reaktion på DNA-skader, kan derefter måles nøjagtigt i flydende medium (se fig.1 for nærmere oplysninger). På denne måde blev der påvist mere end ti nye din-gener, og de fleste af dem blev efterfølgende identificeret.
Kinetik for induktion af β-galactosidase i din::lacZ-fusionsstammer ved hjælp af mitomycin C (MC) . Din::lac-fusionerne blev frembragt ved indsættelse af bakteriofagen Mu d1(Ap, lac) i E. coli-kromosomet. λ::Mu d1(Ap lac)-derivatet blev frembragt ved en indsættelse af Mu d1(Ap lac)-fagen i λ-fagen i E. coli-kromosomet. Symboler: o, ubehandlet fusionsstamme; ●, fusionsstammer plus MC; lexA(Ind-)-derivater af fusionsstammen plus MC; ■, recA(Def)-derivater af fusionsstammen plus MC; ▼, et pKM280-holdigt derivat af den af λ:: Mu d1(Ap lac)-stammen plus MC. Genudskrevet fra med forfatterens tilladelse. To af generne, dinA og dinB blev efterfølgende identificeret som henholdsvis polB (Pol II) og Pol IV .
 (Klik på billedet for at forstørre.)
(Klik på billedet for at forstørre.)
For nylig er der blevet udarbejdet en ny metode til at måle SOS-genekspression og promotoraktivitet af SOS-generne (f.eks, recA, lexA, umuDC) ved at anvende et plasmid med en SOS-promotor, der skal undersøges, fusioneret med reportergenet gfp, der koder for grønt fluorescerende protein (GFP) . Dette gør det muligt at måle promoteraktiviteten af SOS-generne i en enkelt bakteriecelle samt lokalisering og varighed af SOS-induktionen. Det viser sig, at induktion af SOS-generne ikke foregår som en enkelt begivenhed, men følger i flere gentagelige trin, hvis modulation afhænger af den SOS-inducerende dosis, niveauet af skader i DNA’et og UmuD’-proteinakkumulationen. Denne metode åbner en ny mulighed for at måle dynamikken i SOS-responset.
3.2. Ved at søge efter SOS-bokse: Bestemmelse af heterologiindeks (HI)
Fremskridt inden for DNA-sekventering og viden om de karakteristiske elementer i SOS-gensekvenser har gjort det muligt at foretage direkte beregningsmæssige søgninger efter SOS-inducerbare gener. Da 33% af E. coli kromosomale DNA var blevet sekventeret, lokaliserede Lewis et al. ved hjælp af sekvensanalyse og kvantitative DNA-bindingseksperimenter seks nye potentielt LexA-regulerede gener og kaldte dem sosA-F . For to af disse, sosC og sosD, bekræftede forfatterne eksperimentelt, at de binder stærkt til renset LexA-repressor.
Derpå fastslog de ved at sammenligne sekvenserne af SOS-bokse fra 19 din-gener, der var kendt på det tidspunkt (herunder sosA-sosF), at konsensus-sos-bokssekvensen er et perfekt palindrom, TACTG(TA)5CAGTA ; og på grundlag af Bergs og von Hippels teori beregnede de matematisk for hver af SOS-boksene et heterologiindeks (HI). Dette indeks angiver, hvor meget en SOS-boks afviger fra konsensus, og når dets værdi er lav, er genet stærkt undertrykt, og når dets værdi er høj, er det lettere at fjerne undertrykkelsen, når dets værdi er høj. Ved et HI på over 15 binder LexA-repressoren sig ikke til SOS-boxen . HI-værdien er således et mål for den relative styrke af LexA-repressorens binding til en given SOS-boks og er ansvarlig for variationen i derepressionspotentialet.
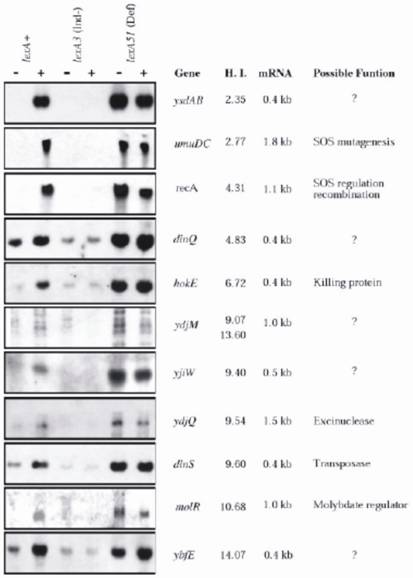
Når DNA-sekvensen for hele E. coli K12-kromosomet er blevet bestemt, lokaliserede Fernandez de Henestrosa et al. ved at søge efter potentielle SOS-bokse, der er forbundet med åbne læserammer, 69 potentielle SOS-bokse med en HI-værdi ≤ 15, herunder alle tidligere kendte og syv nye. De nye gener blev efterfølgende analyseret for deres evne til at blive udtrykt ved MC-behandling, for længden af det udtrykte mRNA og HI-værdierne (fig. 2). Analyserne blev udført i tre isogene E. coli-stammer, der adskiller sig i lexA-allelen: lexA+(wild type) SOS-inducerbar, SOS ikke-inducerbar lexA3(Ind-) og den konstitutivt udtrykte lexA51(Def)-allel. De potentielle funktioner af de nye og gamle gener blev yderligere karakteriseret og diskuteret. Resultaterne bekræftede, at hvert af de nye SOS-box-holdige gener faktisk var et LexA-afhængigt gen, og at dets ekspression kun blev induceret af MC i lexA+-stammen; ellers blev de enten ikke udtrykt (lexA3) eller fuldt ud udtrykt (lexA51), uanset om bakterierne blev MC-behandlet eller ej (se fig. 2 for detaljer).
Fra nukleotidsekvensen af ydjQ-genet (alternative navne b1741 sosD) blev det udledt, at det koder for det 295 aminosyrer lange protein, der deler betydelig homologi med den N -terminale halvdel af UvrC-protein . UvrABC-excinuclease (bestående af UvrA-, UvrB- og UvrC-proteinerne) var kendt for at være involveret i nukleotid-excisonreparation (NER), som fjerner voluminøse addukter eller strukturpåvirkende læsioner (f.eks. pyrimidindimerer, pyrimidin (6-4)-fotoprodukter og abasiske steder) fra modificeret DNA . Det var også kendt, at N-delen af UvrC skærer ssDNA først på 3′-siden af læsionen, og derefter skærer C-delen af UvrC DNA’et på 5′-siden af læsionen. For nylig omdøbte Moolenaar et al. YdjQ-proteinet til Cho (efter UvrC-homolog) og undersøgte dets enzymatiske aktivitet . De bekræftede, at Cho-proteinet kun skærer UvrB-DNA-præincisionskomplekser på 3′-siden af læsionen; nogle læsioner i DNA, som UvrC-proteinet kun meget dårligt skærer, blev dog meget effektivt skæret af Cho-proteinet. Cho-proteinet øger således i høj grad substratområdet i NER-systemets DNA-reparation. Generne uvrA, uvrB og ydjQ (men ikke uvrC) er SOS-inducerede gener.
Nordisk analyse af E. coli-gener, der synes at blive reguleret af LexA. RNA blev ekstraheret fra tre isogene stammer, der adskilte sig i lexA-genet: RW118 (lexA+), RW434 (lexA) og RW542 (lexA51). RNA blev opnået fra ubeskadigede celler (-) og fra celler, der var blevet udsat for mitomycin C (5 µg ml-1) (+) i 30 min. før ekstraktion. De tidligere identificerede LexA-regulerede recA- og umuDC-gener blev anvendt som positive kontroller. Generne er afbildet i henhold til deres stigende heterologiindeks (HI). Størrelsen af mRNA-transkriptet og genernes mulige funktioner er også angivet Se ref. for yderligere oplysninger. (Med venlig høflighed af Blackwell Science).
 (Klik på billedet for at forstørre det.)
(Klik på billedet for at forstørre det.)
3.3. Lokalisering af din gener ved hjælp af mikroarray-teknikker
Mikroarray-teknikken gør det muligt at overvåge et stort antal gener i ét forsøg. Courcelle et al. anvendte mikroarrays, der indeholder forstærkede E coli DNA-kromosomale fragmenter med åbne læserammer fra 4101 gener (95,5 % af det samlede antal) til at måle ekspressionen af alle generne i UV-bestrålede og ikke-bestrålede SOS-inducerbare (lexA+) og ikke-inducerbare lexA1(Ind-) stammer. I den UV-bestrålede lexA+ stamme identificerede forfatterne 17 nye lexA-afhængige SOS-inducerede gener ud over de 26, der var kendt i forvejen; det samlede antal SOS-inducerbare gener i E. coli er derfor sandsynligvis 43. I samme publikation fastslog forfatterne, at ssb-genet, der koder for et ssDNA-bindende protein, ikke er SOS-inducerbart, som man tidligere har troet. De observerede også en række gener, hvis ekspression steg (normalt ikke over to gange) i UV-bestrålede celler, men som ikke blev reguleret af LexA-protein. De bemærkede, at proteintransskriptioner fra mange gener, der ikke var reguleret af LexA, blev reduceret efter UV-stråling, og konkluderede, at disse transskriptioner sandsynligvis enten blev beskadiget eller nedbrudt af UV-stråling. De identificerede også 30 gener med potentielle SOS-bokslignende strukturer, men som ikke var LexA-reguleret.
Mekanisme og specificitet af LexA-repressorbinding til SOS-bokse
Det menes, at sekvenserne af alle potentielle SOS-bokse i E. coli-kromosomet er blevet identificeret. Nogle eksempler på SOS-bokse, der binder (A) eller ikke binder (B og C) LexA-repressoren, sammen med HI-værdier og antallet af mismatches (NM), er vist i tabel 1. NM angiver antallet af positioner i SOS-bokse, der afviger fra et perfekt palindrom. Både antallet og mønsteret af mismatches kan være afgørende for LexA-proteinets specificitet ved binding til hver enkelt SOS-boks. Det ser ud til, at denne hypotese er en god forklaring på specificiteten og den forskellige styrke af LexA-proteinets binding til sekvenser af SOS-bokse. Men dette bør bekræftes.
Det kan ses, at generelt, når SOS-boksene har lave HI-værdier, mellem 2,7 og 12, kan de binde LexA-repressoren (tabel 1A); og når HI er over 16,4 (tabel 1B), er de tilsyneladende ude af stand til at binde LexA-repressoren. I nogle tilfælde (vist i del C) som f.eks. yigN (alternativt navn sosB) og dinJ (sosA)-generne kan SOS-boksene imidlertid ikke binde LexA-repressoren på trods af deres moderate HI-værdier (henholdsvis 9,27 og 7,06) .
Potentielle SOS-bokse for gener, der binder eller ikke binder LexA-repressoren.
| Gen | SOS-boks-sekvens | HI* | NM** | |
|---|---|---|---|---|
| Konsensus | TACTGTATATATATATATACACAGTA | 0 | ||
| A. Gener, hvis SOS-bokse binder LexA og reguleres af LexA-represor | ||||
| recA | TACTGTATGATGAGCATACACAGTA | 4.31 | 1 | |
| umuDC | TACTGTATATATAAAAAAACAGAGTA | 2.77 | 2 | |
| uvrB | AACTGTTTTTTTATCCAGTA | 6.11 | 5 | |
| polB | GACTGTATATATAAAACCACACAGCC | 12.09 | 5 | |
| lexA1 | TGCTGTATATATACTCACACAGCA | 6.34 | 4 | |
| lexA2 | AACTGTATATATACACACACCCAGGG | 8.32 | 6 | |
| B. Gener, hvis potentielle SOS-bokse ikke binder LexA, men som ikke er LexA-reguleret | ||||
| intE | GGCTGCTGCTGAAAAAAATACACAGAA | 16.04 | 7 | |
| ymfI | TTCTGTACCAGAGAAAACAGTT | 15.48 | 8 | |
| ymfM | AGCTGCAGGAGCAGCATGCAGCA | 19.32 | 3 | |
| lit | TGATGACAGAGAGAGTGTCTCCAGTG | 20.32 | 8 | |
| C. Gener, hvis SOS-bokse ikke binder LexA i spate med lav HI-værdi | ||||
| yigN | AACTGGACGTTTGTGTACAGCA | 9,27 | 5 | |
| dinJ | AGCTGAATATATATACAGCA | 7.06 | 3 | |
Potentielle SOS-bokse (sekvens på kodningsstrengen), der binder (A) eller ikke binder (B og C) LexA-repressoren.
HI* angiver heterologiindeks; NM** angiver antallet af mismatches i SOS-bokse, der afviger fra et perfekt palindrom. Manglen på LexA-repressorbinding på trods af en relativt lav HI-værdi (afsnit C) vidner om, at der ikke er nogen direkte korrelation mellem dem . Under alle omstændigheder tyder det på, at HI-værdien ikke kan være den eneste indikator for en SOS-boks’ evne til at binde LexA. Antallet af mismatches i de palindromiske SOS-bokse i hver af afsnittene er ens og er derfor ikke afgørende for LexA-bindingsevnen med SOS-bokserne. Data i del A og C er fra ref. , dem i del B er fra ref. .
Kendetegn ved nogle SOS-inducerede gener
SOS-reaktionsgenerne findes spredt ud over hele E. coli-kromosomet som enkeltgener beliggende i enkeltoperoner. Seks af dem, umuDC (kilden til Pol V), ruvAB (der katalyserer grenvandring i Holliday-strukturer) og ysdAB (med ukendt funktion) er kodet af par af gener, der danner et operon. Generelt er der kun én SOS-box i et operon. Undtagelserne er lexA- og ydjM (b1728)-generne, der hver indeholder to SOS-bokse (adskilt af henholdsvis én og to baser), og recN, der indeholder tre SOS-bokse. Sekvenserne af SOS-boksene i et gen er forskellige . I ydjM’s tilfælde binder to dimeriske LexA-repressorer i samarbejde til hver SOS-boks, og som det skønnes, er de begge funktionelle . Sekvenserne af SOS-boksene i et gen adskiller sig med 2-4 baser. Hvordan og hvorfor de ekstra SOS-bokse i gener opstår, og hvordan de påvirker genekspressionspotentialet, er spørgsmål, der endnu ikke er besvaret.
Den tid, der kræves til derepression af SOS-inducerede gener
Tidsskalaen for derepression af gener og syntese af de SOS-inducerede proteiner varierer for de enkelte gener. De mest hurtigt derepresserede gener (<1 min efter SOS-induktion) omfatter: lexA, der koder for LexA-repressorprotein (hurtigt nedbrudt i SOS-inducerede celler), uvrAB, cho og uvrD, der er involveret i NER-reparation, ruvAB, der deltager i rekombinationel DNA-reparation, polB og dinB, der koder for henholdsvis Pol II og Pol IV , og dinI, hvis produkt hæmmer processeringen af UmuD til UmuD’ . UmuD’-proteinet er nødvendigt for syntesen af det mutagene Pol V (UmuD’2C) . DinI-proteinet forsinker derfor syntesen af Pol V. Ekspressionen af generne recA og recN, der koder for rekombinations- og rekombinationsreparationsproteiner, finder sted 5 min. efter SOS-induktion, mens ekspressionen af sulA (gammelt navn, sfiA) og umuDC finder sted på det seneste tidspunkt af SOS-induktionen . SulA-proteinet er en inhibitor af celledeling, der forårsager filamentøs vækst af cellerne og forlænger den tid, hvor det cellulære DNA kan repareres.
Copy Numbers of din Genes Encoding Proteins
RecA og UvrD hører til de mest hyppigt syntetiserede proteiner. Deres antal på et konstitutivt niveau er henholdsvis omkring 10.000 og 8.000 kopier pr. celle og stiger 10 gange efter SOS-induktion . RecA-proteinet binder sig til ssDNA og beskytter det sandsynligvis mod ukontrolleret ødelæggelse. UvrD, DNA-helicase II, deltager i den dam-instruerede mutHLS-afhængige mismatch-reparation (MMR) , og deltager i UvrABC- og Cho-afhængig (NER) reparation ved at fortrænge det skadesholdige ssDNA fra den reparerede DNA-streng . Antallet af proteinmolekyler, der syntetiseres i uinducerede vs. SOS-inducerede celler, er som følger: 20:250 for UvrA; 250:1000 for UvrB; 40:300 for DNA Pol II, og 250:2500 for DNA Pol IV (11, 34). UmuD-protein udtrykkes, på 180 molekyler pr. uinduceret og på 2400 molekyler pr. manglede funktionel LexA-repressorcelle; der er 200 UmuC-molekyler pr. SOS-induceret celle og ingen Pol V (< 15 molekyler) i uinduceret celle .
Mutagene SOS-inducerede DNA-polymeraser
I E. coli er der ud over den konstitutivt syntetiserede DNA-replikerende Pol III tre potentielt mutagene DNA-polymeraser, hvis syntese er øget (Pol II og Pol IV) eller kun forekommer i SOS-inducerede celler (Pol V). Blandt disse er Pol II den eneste DNA-polymerase, der har en 3′-5′-exonuklease proofreading-aktivitet, og den er den mindst fejlbehæftede; dens rolle omfatter også genoprettelse af nedbrudt DNA ved replikationsgafler . Både pol II og pol IV optræder i de tidlige stadier af SOS-induktionen, og pol V i dens slutfase . Pol V er det mest fejlbehæftede enzym og det vigtigste for mutageniciteten af de SOS-inducerede celler.
I E. coli defekt i umuD(C) induceres næsten ingen mutationer efter UV-stråling, og de mutationer, der induceres ved MMS-behandling, er stærkt reduceret . De vigtigste mutagene læsioner, der dannes i DNA efter UV-stråling, er TT-cis-syn cyclobutan-dimere og CT- eller TT (6-4)-fotoprodukter , mens der efter MMS-behandling findes overvejende 3-methyladenin (3meA), apuriniske steder og 1meA og 3meC (i alkB-mutante celler). Både UV og MMS er ligesom mange andre mutagener SOS-inducerende stoffer, og selv om de skadelige læsioner er forskellige, må det SOS-inducerende signal derfor være fælles; det er almindeligt accepteret, at SOS-inducerende signaler er RecA/ssDNA-filamenter, der dannes på akkumuleret ssDNA i cellerne, når DNA-syntesen er standset. Nogle af de præmutageniske læsioner kræver mutagene DNA-polymeraser for at føre til mutationer, mens andre ikke gør det. Men hvilken SOS-induceret, mutagen DNA-polymerase der kræves, afhænger af typen af læsion.
Slutning
Hypotesen om SOS-respons var forbløffende, frugtbar og inspirerende. Vi indsamlede mange oplysninger om DNA’s metabolisme, udtrykket af de SOS-inducerede gener og deres funktioner. Alligevel er der stadig nye ideer på vej.
Anerkendelser
Jeg er meget taknemmelig over for professor Graham C.Walker, Roger Woodgate og Blackwell Science Publisher for tilladelser til genoptrykning.
Interessekonflikter
Forfatteren har erklæret, at der ikke foreligger nogen interessekonflikter.
1. Weigle JJ. Induktion af mutation i en bakteriel virus. Proc Natl Acad Sci U S A. 1953;39(7):628-636
2. Radman M. Fænomenologi af en inducerbar mutagen DNA-reparationsvej i Escherichia coli: SOS-reparationshypotesen. In: (red.) Sherman S, Miller M, Lawrence C, Tabor WH. Molekylære og miljømæssige aspekter af mutagenese. Springfield IL: Charles C Thomas publisher. 1974:128-142
3. Borek E, Ryan A. Overførsel af bestrålingsudløst induktion i en lysogen organisme. Proc Natl Acad Sci U S A. 1958;44(5):374-347
4. Hertman I, Luria SE. Transduktionsundersøgelser om den rolle, som et rec+ gen spiller i ultraviolet induktion af profage lambda. J Mol Biol. 1967;23(2):117-133
5. Defais M, Fauquet P, Radman M, Errera M. Ultraviolet reaktivering og ultraviolet mutagenese af lambda i forskellige genetiske systemer. Virology. 1971;43(2):495-503
6. Craig R. Funktion af nukleosidtrifosfat og polynukleotid i Escherichia coli recA-proteinrettet spaltning af fage lambda-repressor. J Biol Chem. 1981;256(15):8039-8044
7. Witkin EM. Ultraviolet-induceret mutation og DNA-reparation. Annu Rev Microbiol. 1969;23:487-514
8. Bridges BA. Fejlpræget DNA-reparation og translesion-DNA-syntese II: Den inducerbare SOS-hypotese. DNA Repair (Amst). 2005;4(6):725-739
9. Eller MS, Asarch A, Gilchrest BA. Fotobeskyttelse i den menneskelige hud – et multifacetteret SOS-respons. Phtochem & Photobiol. 2008;84:339-349
10. Clark AJ, Margulies AD. Isolering og karakterisering af rekombinationsdeficiente mutanter af Escherichia coli K12. Proc Natl Acad Sci U S A. 1965;53:451-459
11. Kuzminov A. Rekombinationel reparation af DNA-skader i Escherichia coli og bakteriofagen lambda. Microbiol Mol Biol Rev. 1999;63(4):751-813
12. Horii T, Ogawa T, Nakatani T, Hase T, Matsubara H, Ogawa H. Regulering af SOS-funktioner: oprensning af E. coli LexA-protein og bestemmelse af dets specifikke sted, der spaltes af RecA-proteinet. Cell. 1981;27(3 Pt 2):515-522
13. Little JW, Mount DW. SOS-reguleringssystemet i Escherichia coli. Cell. 1982;29(1):11-22
14. Walker GC. Mutagenese og inducerbare reaktioner på skader på desoxyribonukleinsyre i Escherichia coli. Microbiol Rev. 1984;48(1):60-93
15. Arenson TA, Tsodikov OV, Cox MM. Kvantitativ analyse af kinetikken af endeafhængig disassemblering af RecA-filamenter fra ssDNA. J Mol Biol. 1999;288(3):391-401
16. Schlacher K, Pham P, Cox MM, Goodman MF. Roller af DNA-polymerase V og RecA-protein i SOS-skadeinduceret mutation. Chem Rev. 2006;106(2):406-419
17. Lewis LK, Harlow GR, Gregg-Jolly LA, Mount DW. Identifikation af højaffinitetsbindingssteder for LexA, som definerer nye DNA-skadeinducerbare gener i Escherichia coli. J Mol Biol. 1994;241(4):507-523
18. Thliveris AT, Little JW, Mount DW. Repression af E coli recA-genet kræver mindst to LexA-proteinmonomerer. Biochimie. 1991;73(4):449-456
19. Burckhardt SE, Woodgate R, Scheuermann RH, Echols H. UmuD mutageneseprotein fra Escherichia coli: overproduktion, oprensning og spaltning af RecA. Proc Natl Acad Sci U S A. 1988;85(6):1811-1815
20. Shinagawa H, Iwasaki H, Kato T, Nakata A. RecA-proteinafhængig spaltning af UmuD-protein og SOS-mutagenese. Proc Natl Acad Sci USA. 1988;85(6):1806-1810
21. Tang M, Shen X, Frank EG, O’Donnell M, Woodgate R, Goodman MF. UmuD'(2)C is an error-prone DNA polymerase, Escherichia coli pol V. Proc Natl Acad Sci U S A. 1999;96(16):8919-8924
22. Kenyon CJ, Walker GC. DNA-skadelige stoffer stimulerer genekspression på specifikke loci i Escherichia coli. Proc Natl Acad Sci U S A. 1980;77(5):2819-2823
23. Casadaban MJ, Cohen SN. Laktosegener fusioneret til eksogene promotorer i ét trin ved hjælp af en Mu-lac-bakteriofag: in vivo-sonde for transkriptionelle kontrolsekvenser. Proc Natl Acad Sci U S A. 1979;76(9):4530-4533
24. Bonner CA, Hays S, McEntee K, Goodman MF. DNA-polymerase II er kodet af det DNA-skadeinducerbare dinA-gen fra Escherichia coli. Proc Natl Acad Sci USA. 1990;87:7663-7667
25. Wagner J, Gruz P, Kim SR, Yamada M, Matsui K, Fuchs RPP, Nohmi T. DinB-genet koder for en ny E. coli DNA-polymerase, DNA pol IV, der er involveret i mutagenese. Mol Cell. 1999;4(2):281-286
26. Friedman N, Vardi S, Ronen S, Alin M, Stavans J. Præcis tidsmæssig modulation i responsen fra SOS DNA-reparationsnetværket i individuelle bakterier. PLoS Biol. 2005;3(7):e238
27. Krishna S, Maslov S, Sneppen K. UV-induceret mutagenese i Escherichia coli SOS-respons: en kvantitativ model. PLoS Comput Biol. 2007;3(3):e41
28. Fernandez de Henestrosa AR, Ogi T, Aoyagi S, Chafin D, Hayes JJ, Ohmori H, Woodgate R. Identifikation af yderligere gener tilhørende LexA-regulonen i Escherichia coli. Mol Microbiol. 2000;35(6):1560-1572
29. Selby CP, Sancar A. Mekanismer for transkription-reparationskobling og nedgang i mutationsfrekvensen. Microbiol Rev. 1994;58(3):317-29
30. Moolenaar GF, van Rossum-Fikkert S, van Kesteren M, Goosen N. Cho, a second endonuclease involved in Escherichia coli nucleotide excision repair. Proc Natl Acad Sci U S A. 2002;99(3):1467-1472
31. Courcelle J, Khodursky A, Peter B, Brown PO, Hanawalt PC. Sammenlignende genekspressionsprofiler efter UV-eksponering i Escherichia coli af vild type og SOS-deficient Escherichia coli. Genetics. 2001;158(1):41-64
32. Yasuda T, Morimatsu K, Horii T, Nagata T, Ohmori H. Inhibition af Escherichia coli RecA coprotease af Din I. EMBO J. 1998;17(11):3207-3216
33. Cooper DL, Lahue R.S., Modrich P. Mismatch-reparation i forbindelse med replikationstrofasthed, genetisk rekombination og kræftbiologi. Annu Rev Biochem. 1996;65:101-133
34. Kim S.-R, Matsui K, Yamada M, Gruz P, Nohmi T. Rollerne af kromosomale din-gener, der koder for DNA pol IV i målrettet og ikke-målrettet mutagenese i Escherichia coli. Mol Cell. 1999;4(2):281-286
35. Woodgate R, Ennis DG. Niveauer af kromosomalt kodede Umu-proteiner og krav til in vivo UmuD-spaltning. Mol Gen Genet. 1991;229(1):10-16
36. Rangarajan S, Woodgate R, Goodman MF. En fænotype for enigmatisk DNA-polymerase II i replikationsgenstart i UV-strålede Escherichia coli. Proc Natl Acad Sci USA. 1999;96(16):9224-9229
37. Kato T, Shinoura Y. Isolering og karakterisering af mutanter af Escherichia coli, der er mangelfulde med hensyn til induktion af mutationer ved ultraviolet lys. Mol Gen Genet. 1977;156(2):121-131
38. Sledziewska-Gojska E, Janion C. Alternative veje for methylmethanesulfonat-induceret mutagenese i Escherichia coli. Mol Gen Genet. 1989;216(1):126-31
39. Nieminuszczy J, Sikora A, Wrzesinski M, Janion C, Grzesiuk E. AlkB dioxygenase i forebyggelsen af MMS-induceret mutagenese i Escherichia coli: effekt af Pol V- og AlkA-proteiner. DNA Repair (Amst). 2006;5(2):181-188
40. Napolitano R, Janel-Bintz R, Wagner J, Fuchs RP. Alle tre SOS-inducerbare DNA-polymeraser (Pol II, Pol IV og Pol V) er involveret i induceret mutagenese. EMBO J. 2000;19(22):6259-6265
41. Fuchs RP, Fujii S, Wagner J. Egenskaber og funktioner hos Escherichia coli: Pol IV og Pol V. Adv Protein Chem. 2004;69:229-264
42. Foster PL. Stressinduceret mutagenese i bakterier. Crit Rev Biochem Molec Biol. 2007;42:373-397
43. Iwasaki H, Nakata A, Walker GC, Shinagawa H. Escherichia coli polB-genet, som koder for DNA-polymerase II, er reguleret af SOS-systemet. J Bacteriol. 1990;172(11):6268-6273